# Podsumowanie
Podsumowanie zajęć. Zadania z zajęć są na tym repozytorium. [Link](http://wolynski.home.amu.edu.pl/E4BC1/index.html) do strony z wykładami. DSTTLI Hasło: E4BC1
---
## LAB 1
Zakres:
- wstęp do języka R
- wykład 1 na stronie
### R
Lista:
```r
# wektory
rep(TRUE, 3)
seq(1, 20, by=1)
order(zad6, decreasing = TRUE)]
# pętle
for(i in 1:length(zad5)){}
while (licznik <= length(x)){}
repeat {
if (licznik > length(x)) {
break
}
}
# funkcja, pakiety
minmax <- function(x){}
install.packages("schoolmath")
library(schoolmath)
```
### Zagadnienia


---
## LAB 2
Zagadnienia:
- ciąg dalszy wprowadzenie do R
- wykład 1 na stronie
### R
Lista:
```r
# ładowanie danych
dane <- read.table("dane1.csv", header = TRUE, sep = ";")
load(url("http://ls.home.amu.edu.pl/data_sets/Centrala.RData"))
ankieta <- read.table("http://ls.home.amu.edu.pl/data_sets/ankieta.txt", header = TRUE)
computers <- read.csv("http://pp98647.home.amu.edu.pl/wp-content/uploads/2021/06/computers.csv")
```
### Zagadnienia
Lista:
- **Wektor** musi zawierać takie same typy, **lista** może różne.
- **Macierze**, ogólniej to są **tablice** reprezentowane przez wektor atomowy
- **Czynniki**: dla ("f", "p", "f") zwraca "f", "p"
- **Ramki danych** to jak w excelu arkusze
---
## LAB 3
Zagadnienia:
- **Statystka opisowa** - zaprezentowanie cechy X na próbce za pomocą tabeli, wykresu
- Wykład 2 na stronie
### R
```r
# rozkład empiryczny
ankieta <- read.table("http://ls.home.amu.edu.pl/data_sets/ankieta.txt", header = TRUE)
empiryczny <- data.frame(cbind(liczebnosc = table(ankieta$wynik),
procent = prop.table(table(ankieta$wynik))))
# wykres ramkowy
barplot(table(ankieta$wynik),
xlab = "Odpowiedzi", ylab = "Odpowiedzi",
main = "Rozkład empiryczny zmiennej wynik")
# inne
install.packages("e1071")
library(e1071)
skewness(x)
kurtosis(x)
```
### Zagadnienia
Lista:
- **Miara asymetrii rozkładu** - w którą stronę - prawo/lewo, zmienna się rozkłada.
- zero to symetryczny
- dodatnie to prawostronnie asymetryczny - lewa część jest większa
- ujemna to lewostronnie asymetryczna - prawa część jest większa

- **Kurtoza** - miara skupienia wartości wokół średniej. Porównuje rozkład empiryczny z rozkładem normalnym.
- Większa niż 0, im większa wartość tym bardziej wartości skupione wokół średniej
- Dla rozkładu normalnego = 0
- Dla ujemnych (min -2) wykres jest bardziej spłaszczony niż rozkłąd normalny

- **Odchylenie standardowe** - intuicyjnie rzecz ujmując, odchylenie standardowe mówi, jak szeroko wartości jakiejś wielkości (na przykład wieku, inflacji, kursu walutowego) są rozrzucone wokół jej średniej.
Im mniejsza wartość odchylenia tym obserwacje są bardziej skupione wokół średniej. Odchylenie standardowe z próby ma trochę inny wzór [link](https://pl.wikipedia.org/wiki/Odchylenie_standardowe#Odchylenie_standardowe_z_próby)
- **Współczynnik zmienności** - podaje się w procentach, jest to relacja odchylenia standardowego ze średnią. Mówi nam jak bardzo wartości odbiegają od siebie. Dzięki temu ze jest w procentach mozemy porównywać rózne rozkłady.
- [Przykład](https://pl.wikipedia.org/wiki/Współczynnik_zmienności)
- **Funkcja gęstości** - nieujemna funkcja rzeczywista, określona dla rozkładu prawdopodobieństwa, taka że całka z tej funkcji, obliczona w odpowiednich granicach, jest równa prawdopodobieństwu wystąpienia danego zdarzenia losowego.
- **Histogram** – składa się z szeregu prostokątów umieszczonych na osi współrzędnych. Prostokąty te są z jednej strony wyznaczone przez przedziały klasowe wartości cechy, natomiast ich wysokość jest określona przez liczebności (lub częstości, ewentualnie gęstość prawdopodobieństwa) elementów wpadających do określonego przedziału klasowego.
- **Kwantyl** rzędu p to taka zmienna dla której prawdopodobieństwo wystąpienia od 0 do tej zmiennej jest równe p.
Kwantyl rzędu 1/2 to inaczej mediana. Kwantyle rzędu 1/4, 2/4, 3/4 są inaczej nazywane kwartylami.
- pierwszy kwartyl (notacja: Q1) = dolny kwartyl = kwantyl rzędu 1/4 = 25% obserwacji jest położonych poniżej
- drugi kwartyl (notacja: Q2) = mediana = kwantyl rzędu 1/2 = dzieli zbiór obserwacji na połowę
- trzeci kwartyl (notacja: Q3) = górny kwartyl = kwantyl rzędu 3/4 = dzieli zbiór obserwacji na dwie części odpowiednio po 75% położonych poniżej tego kwartyla i 25% położonych powyżej

- **Wykres ramkowy**


- **Rozkład empiryczny** – uzyskany na podstawie badania statystycznego opis wartości przyjmowanych przez cechę statystyczną w próbie przy pomocy częstości ich występowania.
- Statystki opisowe - rodzaje

---
## LAB 4
Zagadnienia:
- rozkłady statystyczne
- wykład 3 i 4 na stronie
### R
```r
# odchylenie standardowe dla próby to musimy dodatkowo pomnozyc przez ten pierwiastek na koncu!!!
a_est_mm <- mean(czas_oczek_tramwaj) - sqrt(3) * sd(czas_oczek_tramwaj) * sqrt((length(czas_oczek_tramwaj) - 1) / (length(czas_oczek_tramwaj)))
barplot(counts,
xlab = "Liczba zgloszen", ylab = "Prawdopodobienstwo",
main = "Rozklady empiryczny i teoretyczny liczby zgloszen",
col = c("red", "blue"), legend = rownames(counts), beside = TRUE)
#kwanty-kwantyl
qqplot(rpois(length(Centrala$Liczba), lambda = lambda_est), Centrala$Liczba,
xlab = "Kwantyle teoretyczne", ylab = "Kwantyle empiryczne",
main = "Wykres kwantyl-kwantyl dla liczby zgloszen")
qqline(Centrala$Liczba, distribution = function(probs) { qpois(probs, lambda = lambda_est) })
# odchylenie standardowe dla próby to musimy dodatkowo pomnozyc przez ten pierwiastek na koncu!!!
a_est_mm <- mean(czas_oczek_tramwaj) -
sqrt(3) * sd(czas_oczek_tramwaj) * sqrt((length(czas_oczek_tramwaj) - 1) / (length(czas_oczek_tramwaj)))
b_est_mm<- mean(czas_oczek_tramwaj) +
sqrt(3) * sd(czas_oczek_tramwaj) * sqrt((length(czas_oczek_tramwaj) - 1) / (length(czas_oczek_tramwaj)))
# metoda największej warygodności
a_est <- min(czas_oczek_tramwaj)
b_est <- max(czas_oczek_tramwaj)
# metoda najwiekszej warygodnosci
curve(dunif(x, a_est, b_est),
add = TRUE, col = "blue", lwd = 2)
#metoda momentów
curve(dunif(x, a_est_mm, b_est_mm),
add = TRUE, col = "green", lwd = 2)
# bootstrap
library(boot)
dane <- rnorm(100)
meanboot <- function(x,i)mean(x[i])
bmean=boot(dane,meanboot,1000)
hist(bmean$t-mean(dane),prob=T,main='')
curve(dnorm(x,0,1/sqrt(length(dane))),add=T,col='red')
# monte carlo
dane <- rnorm(100)
mcmean <- vector('numeric',1000)
for(i in 1:1000) mcmean[i] <- mean(rnorm(100))
hist(mcmean,prob=T,main='')
curve(dnorm(x,0,0.1),add=T,col='red')
```
### Rozkłady statystyczne
Jeżeli próbka jest reprezentatywna, to stanowi ona podstawę do wnioskowania o populacji z której pochodzi. Wnioskowanie takie wymaga zbudowania modelu “zachowania się” zmiennej (cechy) X w populacji. Budowa modelu polega na przyjęciu założenia o rozkładzie (teoretycznym) zmiennej X w populacji oraz traktowaniu obserwacji jako wartości tej zmiennej.
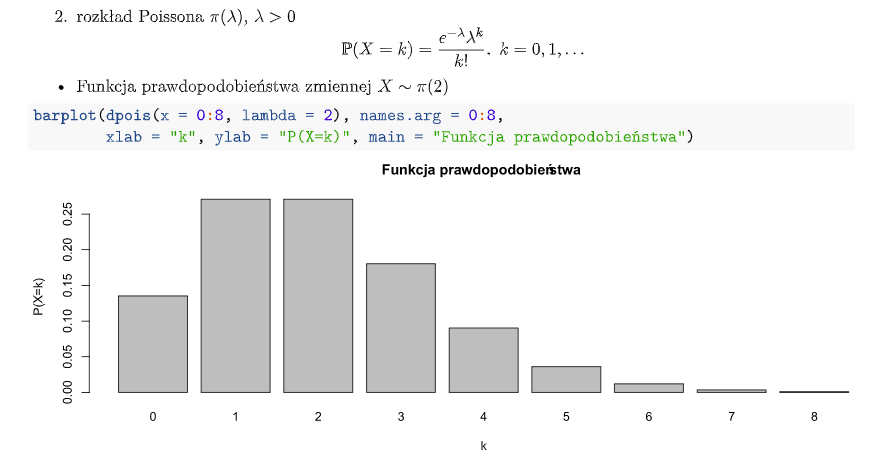
W wykresach na dole to wartość cechy a wysokość słupka to prawdopodobieństwo wystąpienia tej wartości.
- Rozkład Dwumianowy:


- Rozkład Poissona:
- Rozkład Jednostajny:
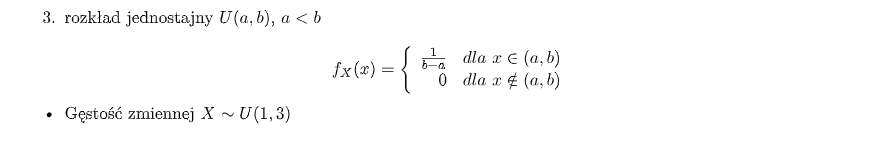

- Rozkład Normalny:

- Rozkład Wykładniczy:


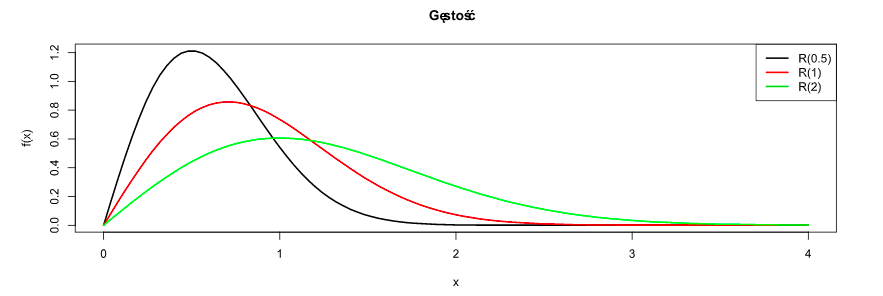
- Rozkład Rayleigha:

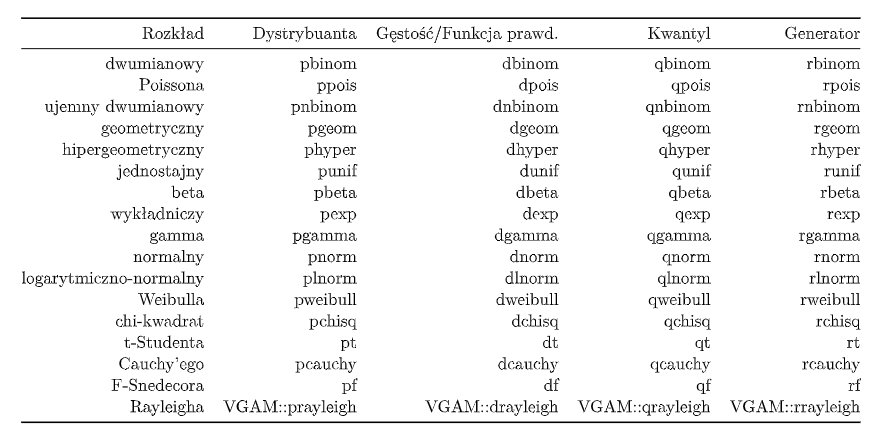
- Inne rozkłady:
### Zagadnienia
- Wykres kwantyl-kwantyl - służy do porównania dwóch rozkładów na podstawie kwantyli. Może służyć do porównania wartości estymowanych z rzeczywistymi. Punkt (x,y) odpowiada jednemu kwantylowi drugiego rodzaju - współrzędna y względem kwantyla tego samego rzędu pierwszego rozkładu - współrzędna x.

- Empiryczne - wynikające z doświadczenia
- Próba statystyczna – zbiór obserwacji statystycznych wybranych (zwykle wylosowanych) z populacji.
### Estymacja
- Estymator - statystyka (funkcja mierzalna określona na przestrzeni statystycznej) służąca do szacowania wartości parametru rozkładu.
- Estymator nieobciążony - wartość oczekiwana rozkładu estymatora jest równa wartości szacowanego parametru.
- Moment - moment zwykły rzędu k zmiennej losowej to wartość oczekiwana k-tej potęgi tej zmiennej.
- zmienna losowa to funkcja prawdopodobieństwa
- wartość oczekiwana to wartość określająca spodziewany wynik doświadczenia losowego. Dobrym estymatorem wartości oczekiwanej jest średnia.

- Metody wyznaczania estymatorów:
- Metoda momentów. Zwykle momenty uk są funkcjami parametrów. Tworzymy układ równań uk = estymator momentu


- Metoda największej wiarogodności

- Metoda Monte Carlo - losowanie, porównywanie


- Metoda bootstrapowa - mamy jakąś próbę z n obeserwacjami i z tej próby losujemy elementy - uzyskujemy próbkę bootstrapową. Powtarzając ten proces otrzymujemy ciąg próbek i odpowiadających jej wartości statystyki. Dzięki tej metodzie, wyniki testów parametrycznych i analiz opartych o modele liniowe są bardziej precyzyjne. Metoda szacowania (estymacji) wyników poprzez wielokrotne losowanie ze zwracaniem z próby. Przydatna gdy nie znamy typu rozkładu.

- Rozkłady estymatorów
- chi-kwadrat - rozkład zmiennej losowej, która jest sumą k kwadratów niezależnych zmiennych losowych o standardowym rozkładzie normalnym.

- Model wykładniczy

- Model normalny

---
## LAB 5
Zagadnienia:
- przedziały ufności
- wykład 5 na stronie
### R
```r
# klasyczne przedziały ufności
library(EnvStats)
epois(Centrala$Liczba,
method = "mle/mme/mvue",
ci = TRUE, ci.type = "two-sided", conf.level = 0.95,
ci.method = "exact")$interval$limits
eexp(Czas,ci=T)
# klasyczne przedziały ufności
load("Awarie.RData")
attach(Awarie)
m <- mean(Czas)
n <- length(Czas)
a <- 0.05
# chi-kwadrat
L <- qchisq(a/2,2*n)/(2*n*m)
R <- qchisq(1-(a/2),2*n)/(2*n*m)
# bootstrapowe przedziały ufności
library(boot)
load("Awarie.RData")
attach(Awarie)
lambdaboot <- function(x,i) 1/mean(x[i])
blambda <- boot(Czas,lambdaboot,1000)
boot.ci(blambda,conf=0.95,type='perc')
```
### Zagadnienia
- Estymacja przedziałowa - np jakieś urządzenie moze działać na pewnym przedziale wartości.
- Chcemy "złapać" jakąś wartość w przedział. Jest to lepsze niz próba oszacowania dokładnej wartości.
Jeżeli konstruujemy jakiś przedział z poziomem ufności 0,95 to na 100 prób w 95 nasz parametr jest w przedziale.
- Na podstawie funkcji centralnej mozemy stworzyć przedziały ufności. Funkcję centralną bierzemy z tabelki.
Podstawiamy funkcję centralną do prawdopodobieństwa oraz 1-a, wsadzamy parametr pomiędzy funkcję i wyliczamy a i b. a i b to przedział ufności. Jest przykład w pdfie w labach 5.

- Rozkład t-Studenta - kolejny typ rozkładu. Podobny to rozkładu normalnego. Rozkład t studenta stosujemy tylko w sytuacji, gdy odchylenie standardowe populacji jest nieznane, a rozmiar próby(ilość obserwacji) jest mniejsza niż 30. W przypadku gdy rozmiar próby jest większy lub równy 30 wtedy zamiast brać rozkład t bierzemy rozkład normalny. Wynika to z faktu, że rozkład t studenta dla 𝑛≥30 jest bardzo podobny do rozkładu normalnego. Dla n < 30 rozkład studenta jest „szerszy”, tzn. bardziej prawdopodobne są wartości mocno odbiegające od średniej niż w przypadku rozkładu normalnego.

- Dodatkowe

- Bootstrapowe przedziały ufności - po prostu przedział ufności z próbki bootstrapowej. (Z niewielkiej próby tworzymy losując ze zwracaniem zestaw wielu prób)

---
## LAB 6
Zagadnienia:
- testy statystyczne, testowanie hipotez statystycznych
- testy t-studenta
- wykład 6 i 7 na stronie
### R
```r
x <- c(78.2, 78.5, 75.6, 78.5, 78.5, 77.4, 76.6)
y <- c(76.1, 75.2, 75.8, 77.3, 77.3, 77.0, 74.4, 76.2, 73.5, 77.4)
boxplot(x, y)
shapiro.test(x)$p.value
qqnorm(x)
qqline(x)
shapiro.test(y)$p.value
qqnorm(y)
qqline(y)
var(x)
var(y)
var.test(x, y, alternative = "less")$p.value
#test t-studenta dla jednej próby
load("Hamulce.RData")
attach(Hamulce)
mean(Wynik)
t.test(Wynik,mu=18.6,alternative='less')
```
## Testy statystyczne
- Testujemy czy wartość parametru jest istotnie różna od zadanej wartości. Musimy podać hipotezę alternatywną - działanie które podejmujemy jeśli hipoteza zerowa jest fałszywa.
- Obszary krytyczne

- Błędy pierwszego i drugiego rodzaju. Przez to możemy podjąć dwie decyzje - "odrzucamy hipotezę zerową" lub "nie ma podstaw do odrzucenia hipotezy zerowej".
- Odrzucamy hipotezę zerową gdy jest ona prawdziwa - błąd I rodzaju.
- Przyjmujemy hipotezę zerową gdy jest ona fałszywa - błąd II rodzaju.
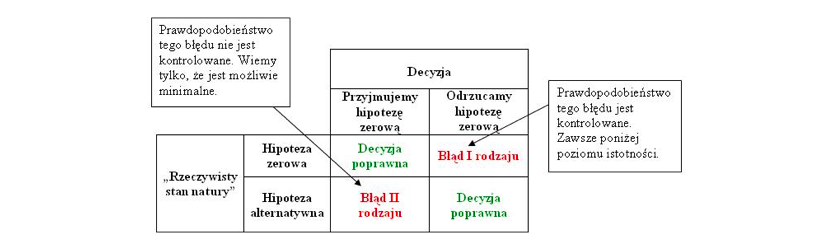
- Wybór wartości krytycznej - Ustalamy poziom istotności testu α i dobieramy wartość krytyczną tak, aby
- prawdopodobieństwo popełnienia błędu I rodzaju było mniejsze lub równe α,
- prawdopodobieństwo popełnienia błędu II rodzaju było minimalne.
- Testy ilorazu wiarogodności

### Zagadnienia
- P-wartość (p-value) to graniczny poziom istotności - najmniejszy, przy którym zaobserwowana wartość statystyki testowej prowadzi do odrzucenia hipotezy zerowej. Im p-wartość jest większa, tym bardziej hipoteza H0 jest prawdziwa. Im mniejsza tym niej prawdopodobna jest hipoteza H0. Wartość p, p-wartość, prawdopodobieństwo testowe. Sposoby obliczania z obszaru:
- Prawostronny obszar krytyczny
- Lewostronny obszar krytyczny
- Dwustronny obszar krytyczny
- Test t Studenta jest metodą statystyczną służącą do porównania dwóch średnich między sobą jeśli znamy liczbę badanych osób, średnią arytmetyczną oraz wartość odchylenia standardowego lub wariancji.
Jest to jeden z mniej skomplikowanych i bardzo często wykorzystywanych testów statystycznych używanych do weryfikacji hipotez. Dzięki niemu możemy dowiedzieć się czy dwie różne średnie są różne niechcący (w wyniku przypadku) czy są różne istotnie statystycznie (np. z uwagi na naszą manipulację eksperymentalna).
Są gotowe wzory do których podstawiamy wartości w zalezności od rodzaju próby. **Przykład w pdf w labach 6 - dla jednej próby lub dla dwóch wzory są na stronie**.
- Założenie normalności rozkładów błędów możemy (ewentualnie) zastąpić założeniem mówiącym o dysponowaniu dużą próbą, tzn.
- Próby niezależne - obserwacje w poszczególnych populacjach (grupach) dokonywane są na różnych jednostkach eksperymentalnych.
- Próby zależne - obserwacje dokonywane są dwukrotnie na tych samych jednostkach eksperymentalnych.
- Test Shapiro-Wilka- hipotezy:
- H0 : Próba pochodzi z populacji o rozkładzie normalnym
- H1 : Próba nie pochodzi z populacji o rozkładzie normalnym.
Hipoteza zerowa tego testu mówi nam o tym, że nasza próba badawcza pochodzi z populacji o normalnym rozkładzie. Jeśli test Shapiro-Wilka osiąga istotność statystyczną (p < 0,05), świadczy to o rozkładzie oddalonym od krzywej Gaussa. W przypadku tego testu najczęściej chcemy otrzymać wartości nieistotne statystyczne (p > 0,05), ponieważ świadczą one o zgodności rozkładu zmiennej z rozkładem normalnym.
- Var.test (test F dla dwóch wariancji) - wariancja - Intuicyjnie utożsamiana ze zróżnicowaniem zbiorowości. Wg dokumentacji jest to test pozwalający porównać wariancje z dwóch rozkładów normalnych.
---
## LAB 7
Zagadnienia:
- analiza wariancji ANOVA - w skróce badanie zależności między obserwacjami
- wykład 8 na stronie
### R
```r
# założenia
rests <- lm(number ~ context, data = dane)$residuals
shapiro.test(rests)$p.value
qqnorm(rests)
qqline(rests)
bartlett.test(number ~ context, data = dane)$p.value
fligner.test(number ~ context, data = dane)$p.value
library(car)
leveneTest(number ~ context, data = dane)[[3]][1]
leveneTest(number ~ context, data = dane, center = "mean")[[3]][1]
# jednoczynnikowa ANOVA
summary(aov(number ~ context, data = dane))
# testy post-hoc
pairwise.t.test(number, context, data = dane)
TukeyHSD(model_aov)$context
plot(TukeyHSD(model_aov))
install.packages("agricolae")
library(agricolae)
HSD.test(model_aov, "context", console = TRUE)
SNK.test(model_aov, "context", console = TRUE)
LSD.test(model_aov, "context", p.adj = "holm", console = TRUE)
scheffe.test(model_aov, "context", console = TRUE)
# kontrasty
C1 <- c(-3, 2, 2, -3, 2)
C2 <- c(0, -1, -1, 0, 2)
C3 <- c(0, 1, -1, 0, 0)
C4 <- c(1, 0, 0, -1, 0)
con <- cbind(C1, C2, C3, C4)
contrasts(dane$context) <- con
model_aov_2 <- aov(number ~ context, data = dane)
summary(model_aov_2, split = list(context = list('C1' = 1, 'C2' = 2, 'C3' = 3, 'C4' = 4)))
```
### Wstęp
- Analiza wariancji ANOVA - metoda statystyczna służąca do badania obserwacji, które zależą od jednego lub wielu działających równocześnie czynników. Metoda ta wyjaśnia, z jakim prawdopodobieństwem wyodrębnione czynniki mogą być powodem różnic między obserwowanymi średnimi grupowymi.
- Modele jednoczynnikowe – wpływ każdego czynnika jest rozpatrywany oddzielnie, tą klasą zagadnień zajmuje się jednoczynnikowa analiza wariancji. Chcemy sprawdzić, czy jakiś pojedynczy czynnik ma wpływ na mierzoną zmienną zależną.
- Modele wieloczynnikowe – wpływ różnych czynników jest rozpatrywany łącznie, tą klasą zagadnień zajmuje się wieloczynnikowa analiza wariancji.
### Jednoczynnikowa analiza wariancji
Na test jednoczynnikowej analizy wariancji możemy patrzeć jak na uogólnienie testu t-Studenta dla więcej niż 2 prób niezależnych.

- Wyniki przedstawiamy w postaci tabeli

- Założenia:
- Niezależność obserwacji dla poszczególnych jednostek eksperymentalnych.
- Błędy mają rozkłady normalne z zerową wartością oczekiwaną (brak błędu systematycznego) i jednorodną wariancją. - to możemy sprawdzić **testem Bartletta**
## Testy post hoc
Testy post-hoc (po fakcie) wykonuje się jako kolejny krok analizy wariancji. Znane są również pod nazwą porównań wielokrotnych lub porównań parami. Sama analiza wariancji mówi nam o tym czy różnice w porównywanych średnich występują czy nie. Nie wiemy jednak między którymi grupami zachodzą te różnice. Istotny współczynnik F wskazuje jedynie na słuszność (lub brak słuszności) odrzucenia hipotezy zerowej. Jeśli ją odrzucimy musimy dowiedzieć się czy wszystkie średnie różnią się między sobą czy tylko niektóre. Stąd też nazwa “po fakcie” – wykonujemy je dopiero po sprawdzeniu czy wynik F jest istotny statystycznie. Jeśli nie jest, nie musimy wykonywać testów post-hoc.
- Procedura NIR – Fishera.

- Tukey, test HSD i SNK są pochodnymi Tukeya

- LSD - Multiple comparisons, "Least significant difference" and Adjust P-values. Wynik też grupy.
- Scheffe - method is very general in that all possible contrasts can be tested for significance and confidence intervals can be constructed for the corresponding linear. The test is conservative. Wynik też grupy.
- pairwise.t.test - daje wynik w postaci tabeli jak różnią się od siebie wyniki poszczególnych grup
### Zagadnienia
- bartlett.test, fligner.test oraz leveneTest użyte do sprawdzenia czy wariancje dwóch grup są takie same.

- Układ losowych bloków kompletnych. W celu wyeliminowania niejednorodności jednostek eksperymentalnych możemy pogrupować je w bloki. Po prostu przed testami grupujemy.
- Jednoczynnikowa ANOVA - układ doświadczalny. Na stronie analiza wariancji była wytłumaczona bardziej opisowo - na podstawie analogii do układu doświadczalengo.
- Kontrasty - by można było zdefiniować wybrane hipotezy należy dla każdej średniej przypisać wartość kontrastu.
Wartości kontrastu są tak wybierane by ich sumy dla porównywanych stron były liczbami przeciwnymi, a ich wartość dla średnich nie biorących udziału w analizie wynosi 0!!
Wykonując później test ANOVA - analiza wariancji możemy zobaczyć czy porównywane grupy (wyszczególnione za pomocą kontrastów) różnią się od siebie.
- Ortogonalny - prostopadły
---
## LAB 8
Zagadnienia:
- regresja liniowa
- wykład 9 na stronie - początek
### R
```r
# regresja liniowa
model <- lm(liczba_przypadkow ~ rok, data = data_set)
model$coefficients
plot(data_set, main = "Wykres rozrzutu", pch = 16)
abline(model, col = "red", lwd = 2)
coef(model)
confint(model)
# bez wyrazu wolnego
model <- lm(distance ~ speed - 1, data = braking)
# szczegóły
summary(model)
fitted(model)
residuals(model)
summary(model_1_3)$adj.r.squared # dopasowanie modelu
# przedziały ufności (jakbym usunął to +- 10 to nic się nie zmienia właściwie)
temp_rok <- data.frame(rok = seq(min(data_set$rok) - 10,
max(data_set$rok) + 10,
length = 100))
pred <- stats::predict(model, temp_rok, interval = "prediction")
plot(data_set, main = "Wykres rozrzutu", pch = 16)
abline(model, col = "red", lwd = 2)
lines(temp_rok$rok, pred[, 2], lty = 2, col = "red")
lines(temp_rok$rok, pred[, 3], lty = 2, col = "red")
# predykcja
new_rok <- data.frame(rok = 2003:2007)
(pred_2003_2007 <- stats::predict(model, new_rok, interval = 'prediction'))
plot(data_set, main = "Wykres rozrzutu z predykcją na lata 2003-2007", pch = 16,
xlim = c(1995, 2007), ylim = c(10, 40))
abline(model, col = "red", lwd = 2)
points(2003:2007, pred_2003_2007[, 1], col = "blue", pch = 16)
temp_rok <- data.frame(rok = seq(1994, 2008, length = 100))
pred <- stats::predict(model, temp_rok, interval = "prediction")
lines(temp_rok$rok, pred[, 2], lty = 2, col = "red")
lines(temp_rok$rok, pred[, 3], lty = 2, col = "red")
```
### Regresja
Główną ideą regresji jest przewidywanie, prognozowanie danych dla pewnej zmiennej na podstawie innych zmiennych. Innymi słowy, jaką wartość przyjmie dana zmienna gdy będziemy znali wartość innej zmiennej. Oczywiście, aby móc "poszukiwać" wartości jednej zmiennej na podstawie innej zmiennej musimy za pomocą analizy regresji skonstruować model regresyjny, model, który będzie z założonym błędem statystycznym przewidywał wartość, poziom danej cechy.
Założenia analizy regresji:
- Niezależność obserwacji dla poszczególnych jednostek eksperymentalnych.
- Brak błędu systematycznego.
- Jednakowa i stała wariancja błędów.
- Brak korelacji błędów.
- W procedurach testowych oraz w przypadku wykorzystywania przedziału predykcji, potrzebne jest dodatkowe założenie normalności błędów. Powoduje ono, że brak korelacji błędów oznacza ich niezależność.

- Estymacja funkcji regresji

### Zagadnienia
- Regresja liniowa - jest najprostszym wariantem regresji w statystyce. Zakłada ona, że zależność pomiędzy zmienną objaśnianą a objaśniająca jest zależnością liniową.

- Poziom ufoności - jak często mamy rację. Wyrażane w procentach.
- Reszty - o ile różni się wynik zmierzony od przewidzianego.
- Czasem można usunąć wyraz wolny. Analogicznie jest ze współczynnikiem kierunkowym (wtedy zmienne są niezależne) - wzór na stronie

- Dopasowanie modelu

---
## LAB 9
Zagadnienia:
- regresja wielokrotna i krokowa
- wykład 9 na stronie środek
### R
```r
# regresja wielokrotna
pairs(auto_wna_sel)
model_1 <- lm(price ~ horsepower + city.mpg + peak.rpm + curb.weight + num.of.doors, data = auto_wna)
coef(model_1)
confint(model_1)
summary(model_1)
fitted(model_1)
residuals(model_1)
# regresja krokowa w tył (jeden krok)
step(model_1) # AIC
step(model_1, k = log(nrow(auto_wna))) # BIC
# regresja krokowa w przód (jeden krok)
model_0 <- lm(price ~ 1, data = auto_wna)
step(model_0, direction = "forward", scope = formula(model_1))
# predykcja
new_data <- data.frame(curb.weight = 2823, horsepower = 154)
model_2 <- lm(price ~ curb.weight + horsepower, data = auto_wna)
stats::predict(model_2, new_data, interval = "prediction")
summary(model_2)$adj.r.squared
```
### Zagadnienia
- Regresja wielokrotna - z regresją wielokrotną (wieloraką) mamy do czynienia, jeśli zmianna zależna Y związana jest z większą ilością zmiennych niezależnych.
- Na stronie są wzory z dopasowaniem, pedykcją, odrzuceniem wyrazu wolnego i estymatorami - analogicznie jak do regresji liniowej.

- Regresja krokowa - regresja krokowa jest odmianą analizy regresji (nie tylko regresji liniowej), w której do modelu wprowadzane są jedynie istotne statystycznie zmienne, predyktory, które rzeczywiście „poprawiają” zbudowany model. Możemy odrzucić lub dodać zmienną w każdym kroku.
- Jeśli zaczynamy od stałej i dodajemy zmienne to jest to regresja w przód.
- Możemy też zacząć od wszyskich zmiennych i odejmować - regresja w tył.
- Decydujemy o dodaniu / sprawdzamy co się stanie jak odejmiemy - za pomocą kryterium np test istotności AIC, BIC
- AIC - kryterium wyboru pomiędzy modelami statystycznymi o różnej liczbie predyktorów.

- BIC - w modelowaniu równań strukturalnych jest to jeden ze wskaźników dopasowania modelu. Minimalizujemy wartość wskaźnika.
- step z k=log to regresja krokowa BIC, k=2 to AIC
- L to zmaksymalizowana funkcja prawdopodobieństwa modelu M L=p(x|theta,M)
- n to liczba obserwacji
- k - liczba parametrów

---
## LAB 10
Zagadnienia:
- regresja logistyczna i Poissona
- wykład 9 końcówka
### R
```r
# regresja logistyczna
model_1 <- glm(condition ~ bilirubin + ldh, data = liver_data, family = 'binomial')
summary(model_1)
step(model_1)
# iloraz szans
exp(coef(model_1)[2])
exp(coef(model_1)[3])
# krzywa ROC
install.packages("ROCR")
library(ROCR)
pred_1 <- prediction(model_1$fitted, liver_data$condition)
plot(performance(pred_1, 'tpr', 'fpr'), main = "Model 1")
performance(pred_1, 'auc')@y.values
# predykcja z wykresem
liver_data_new <- data.frame(bilirubin = c(0.9, 2.1, 3.4), ldh = c(100, 200, 300))
(predict_glm <- stats::predict(model_1, liver_data_new, type = 'response'))
model_1_hat <- coef(model_1)[1] + coef(model_1)[2] * liver_data$bilirubin + coef(model_1)[3] * liver_data$ldh
model_1_temp <- seq(min(model_1_hat) - 1, max(model_1_hat) + 2.5, length.out = 100)
condition_temp <- exp(model_1_temp) / (1 + exp(model_1_temp))
plot(model_1_temp, condition_temp, type = "l", xlab = "X beta", ylab = "condition", xlim = c(-6, 9), ylim = c(-0.1, 1.1))
points(model_1_hat, liver_data$condition, pch = 16)
points(coef(model_1)[1] + coef(model_1)[2] * liver_data_new$bilirubin + coef(model_1)[3] * liver_data_new$ldh, predict_glm, pch = 16, col = "red")
# regresja Poissona
install.packages("DAAG")
library(DAAG)
head(moths)
model_1 <- glm(A ~ log(meters), data = moths, family = 'poisson')
# predykcja
data_new <- data.frame(meters = c(3, 20, 100))
(pred_1 <- stats::predict(model_1, data_new, type = "response"))
moths$A_hat <- stats::predict(model_1, type = "response")
moths <- moths[with(moths, order(meters)), ]
plot(log(moths$meters), moths$A_hat,
type = "l", col = "red", lwd = 2,
xlab = "log(moths$meters)", ylab = "A",
ylim = c(0, 40), main = "Model 1")
points(log(moths$meters), moths$A, pch = 16)
points(log(data_new$meters), pred_1, pch = 16, col = "blue", lwd = 4)
```
### Krzywa ROC
W statystyce matematycznej krzywa ROC jest graficzną reprezentacją efektywności modelu predykcyjnego poprzez wykreślenie charakterystyki jakościowej klasyfikatorów binarnych powstałych z modelu przy zastosowaniu wielu różnych punktów odcięcia. Mówiąc inaczej – każdy punkt krzywej ROC odpowiada innej macierzy błędu uzyskanej przez modyfikowanie „cut-off point”. W punkcie (0, 0) model klasyfikuje wszystko jako negative w punkcie (1, 1) model klasyfikuje wszystko jako positive. Pojęcia:
- TPR (True Positive Rate) – określa zdolność klasyfikatora do wykrywania klasy pozytywnej
- Lepiej jest powyżej przekątnej, gorzej poniżej. Przekątna to klasyfikator losowy. Przekątna idzie od początku układu współrzędnych.
- False positive rate = 1-specyficzność
- Punkt równowagi to klasyfikator gdzie czułość = specyficzność. Jest to na przecięciu drugiej przekątnej z wykresem.

- AUC - Interpretacja AUROC (Area Under the ROC) to prawdopodobieństwo, że badany model predykcyjny oceni wyżej (wartość score) losowy element klasy pozytywnej od losowego elementu klasy negatywnej. Jest to dokłądność modelu - dla idealnego AUC=100%.

### Zagadnienia
- Regresja logistyczna – jedna z metod regresji używanych w statystyce w przypadku, gdy zmienna zależna jest na skali dychotomicznej (przyjmuje tylko dwie wartości). Zmienne niezależne w analizie regresji logistycznej mogą przyjmować charakter nominalny, porządkowy, przedziałowy lub ilorazowy. W przypadku zmiennych nominalnych oraz porządkowych następuje ich przekodowanie w liczbę zmiennych zero-jedynkowych taką samą lub o 1 mniejszą niż liczba kategorii w jej definicji.
Zwykle wartości zmiennej objaśnianej wskazują na wystąpienie, lub brak wystąpienia pewnego zdarzenia, które chcemy prognozować. Regresja logistyczna pozwala wówczas na obliczanie prawdopodobieństwa tego zdarzenia (tzw. prawdopodobieństwo sukcesu). Wykres krzywej to sigmoid function


- Rozkład Poissona – dyskretny rozkład prawdopodobieństwa, wyrażający prawdopodobieństwo szeregu wydarzeń mających miejsce w określonym czasie, gdy te wydarzenia występują ze znaną średnią częstotliwością i w sposób niezależny od czasu jaki upłynął od ostatniego zajścia takiego zdarzenia.
- Regresja Poissona - ilość wydarzeń.

- Liczba stopni swobody – liczba niezależnych wyników obserwacji pomniejszona o liczbę związków, które łączą te wyniki ze sobą. Liczbę stopni swobody można utożsamiać z **liczbą niezależnych zmiennych losowych**, które wpływają na wynik. Inną interpretacją liczby stopni swobody może być: liczba obserwacji minus liczba parametrów estymowanych przy pomocy tych obserwacji.
- Regresja nieliniowa

- Iloraz szans - w skrócie OR określa nam stosunek szansy wystąpienia danego zdarzenia w danej grupie do wystąpienia tego samego zdarzenia w innej porównywanej grupie.
---
## LAB 11
Zagadnienia:
- powtórka
### R
```r
# definiuje ułożenie wykresów w RStudio
par(mfrow=c(2,2))
# wykresy gęstości jądrowych przy różnych szerokościach okna
x1<-rexp(30,5)
x2<-rnorm(30,2,2)
x3<-rnorm(30,10,1)
gen<-c(x1,x2,x3)
plot(density(gen,bw=0.1))
plot(density(gen,bw=0.5))
```
---
## LAB 12
Zagadnienia:
- analiza składowych głównych
- wykład 10 na stronie
### R
```r
# współczynnik korelacji -1 ujemnie; 0 brak korelacji; 1 dodatnio
cor.test(USArrests$Rape,USArrests$UrbanPop, method="pearson")
# analiza składowych głównych
(pca_1 <- prcomp(~ Murder + Assault + Rape, data = USArrests, scale = TRUE))
summary(pca_1)
# wykres ładunków - wartość bezwzględna
matplot(abs(pca_1$rotation), type = 'l', lty = 1, lwd = 2,
xlab = 'zmienne', ylab = '|ładunki|', ylim = c(0, 1.05),
xaxt = "n")
axis(1, at = 1:3, labels = rownames(pca_1$rotation))
legend('topleft', legend = c('PC1', 'PC2', 'PC3'), ncol = 3, col = 1:3, lwd = 2)
text(rep(1, 3), abs(pca_1$rotation)[1, ], abs(round(pca_1$rotation[1, ], 2)), pos = 4)
text(rep(2, 3), abs(pca_1$rotation)[2, ], abs(round(pca_1$rotation[2, ], 2)), pos = 1)
text(rep(3, 3), abs(pca_1$rotation)[3, ], abs(round(pca_1$rotation[3, ], 2)), pos = 2)
# wykres osypiska
plot(pca_1)
# średnia
pca_1$sdev^2
mean(pca_1$sdev^2)
# biplot
biplot(pca_1)
# minimalne drzewo
install.packages("ape")
library(ape)
plot(mst(dist(scale(USArrests[, -3]))), x1 = pca_1$x[, 1], x2 = pca_1$x[, 2])
```
### Zagadnienia
- Analiza składowych głównych - jest techniką redukcji wymiaru. Jej celem jest znalezienie niewielkiej liczby składowych głównych, które wyjaśniają w maksymalnym stopniu całkowitą wariancję z próby p zmiennych pierwotnych. S - to estymator nieobciążony.

- Ładunki - dokładniejszą interpretację składowych można uzyskać poprzez wyznaczenie tzw. macierzy ładunków czynnikowych (które są współczynnikami korelacji między i-tą zmienną i j-tą składową).

- Ładunki czynnikowe - podobnie jak współczynniki zawarte w wektorze własnym, odzwierciedlają wpływ poszczególnych zmiennych na daną składową główną.
- Wykres osypiska - wykres wariancji poszczególnych składowych głównych. Możemy odrzucić te dla których wartość jest mniejsza od 80% lub gdy wartości własne skłądowej głównej są mniejsze od średniej

- Biplot

- Można stworzyć minimalne drzewo rozpinające - graf którego wierzchołkami są obserwacje, dwa punkty są połączone jedną ścieżką, a suma krwędzi jest minimalna. Punkty połączone krawędziami powinny być blisko siebie
- Model wielowymiarowy.

- Estymatory

- Kreska nad zmienną oznacza zazwyczaj średnią (przynajmniej na wikipedii)
---
## LAB 13
Zagadnienia:
- analiza korelacji
- wykład 11 na stronie
### R
```r
# test założeń
shapiro.test(mtcars$wt)$p.value
# pearson
cor.test(mtcars$mpg, mtcars$wt, method = "pearson")$p.value
cor.test(mtcars$mpg, mtcars$wt, method = "pearson")$est
# kendall
cor.test(mtcars$mpg, mtcars$wt, method = "kendall")$p.value
cor.test(mtcars$mpg, mtcars$wt, method = "kendall")$est
# spearman
cor.test(mtcars$mpg, mtcars$wt, method = "spearman")$p.value
cor.test(mtcars$mpg, mtcars$wt, method = "spearman")$est
# inne
chisq.test(Efekt,Metoda)
cor(Girth,Volume)
```
### Zagadnienia
- Analiza zależności cech - jeśli badaniu statystycznemu podlega jednocześnie wiele cech, to jednym z podstawowych zagadnień staje się analiza zależności pomiędzy nimi. Do wykrycia zależności pomocne są odpowiednie wykresy, współczynniki mierzące jej siłę oraz testy badające jej istotność.
- Tabela dwudzielcza.

- Test niezależności w tablicy dwudzielczej - po prostu wszyskie możliwe kombinacje są niezależne - na stronie jest rozpisany wzór.

- Test istotności dla współczynnika korelacji - na stronie.

- Korelacja Pearsona - służy do sprawdzenia czy dwie zmienne ilościowe są powiązane ze sobą związkiem liniowym. -1 = odwrotnie liniowa, 0 = brak korelacji, 1 = dodatnio liniowa.
- Korelacja Tau Kendalla - statystyka będąca jedną z miar monotonicznej zależności dwóch zmiennych losowych. -1 = każda maleje przy wzroście drugiej, 0 = brak korelacji, 1 = każda ze zmiannych rośnie przy wzroście drugiej.
- Korelacja rang Spearmana - ich interpretacja jest podobna do klasycznego współczynnika korelacji Pearsona, z jednym zastrzeżeniem: w odróżnieniu od współczynnika Pearsona, który mierzy liniową zależność między zmiennymi, a wszelkie inne związki traktuje jak zaburzone zależności liniowe, korelacja rangowa pokazuje dowolną monotoniczną zależność (także nieliniową).
---
## LAB 14
Zagadnienia:
- klasyfikacja
- wykład 12 na stronie (jest jeszcze więcej wywołań w R)
### R
```r
# liniowa dyskryminacja
library(MASS)
model_lda <- lda(V14 ~ V1 + V2 + V3, data = wina)
# błąd klasyfikacji metodą sprawdzania krzyżowego v=10
library(MASS)
v <- 10
blad_i <- numeric(v)
krok <- floor(nrow(iris) / v)
permutacja <- sample(1:nrow(iris))
temp <- 0
for (i in 1:v) {
if (i != v) {
obs_temp <- permutacja[(temp + 1):(i * krok)]
temp <- i * krok
} else {
obs_temp <- permutacja[(temp + 1):nrow(iris)]
}
model_lda_i <- lda(Species ~ ., data = iris[-obs_temp, ])
pred_i <- stats::predict(model_lda_i, iris[obs_temp, ])$class
blad_i[i] <- sum(pred_i != iris$Species[obs_temp])
}
sum(blad_i) / nrow(iris)
# bootstrap
n_boot <- 100
temp_boot <- numeric(n_boot)
set.seed(1234)
for (i in 1:n_boot) {
numery <- sample(1:nrow(iris), replace = TRUE)
model_lda_i <- lda(Species ~ ., data = iris[numery, ])
temp_boot[i] <- mean(stats::predict(model_lda_i, iris[-numery, ])$class != iris[-numery, ]$Species)
}
mean(temp_boot)
# tablica kontynencji (krzyżowa)
head(stats::predict(model_lda)$posterior)
head(stats::predict(model_lda)$class)
(conf_matrix <- table(stats::predict(model_lda)$class, wina$V14))
# błąd metodą ponownego podstawiania
(1 - sum(diag(conf_matrix)) / nrow(wina))
# klasyfikacja
new_data <- data.frame(V1 = c(13.64, 13.94, 13.08, 12.29),
V2 = c(3.1, 1.73, 3.9, 3.17),
V3 = c(2.56, 2.27, 2.36, 2.21))
stats::predict(model_lda, new_data)
# gotowy bootstrap
library(caret)
ctrl_boot <- trainControl(method = 'boot',
number = 100,
search = 'grid')
ctrl_loo <- trainControl(method = 'LOOCV',
search = 'grid')
ctrl_10CV <- trainControl(method = "repeatedcv",
number = 10,
repeats = 10)
train(V14 ~ V1 + V2 + V3, data = wina, method = 'lda', trControl = ctrl_boot)
train(V14 ~ V1 + V2 + V3, data = wina, method = 'lda', trControl = ctrl_loo)
train(V14 ~ V1 + V2 + V3, data = wina, method = 'lda', trControl = ctrl_10CV)
```
### Zagadnienia
- Klasyfikacja - w zagadnieniach klasyfikacyjnych poszukujemy reguły (klasyfikatora) (zazwyczaj oznaczanego literą d) pozwalającego na przyporządkowanie obiektu ( opisanego przez p wymiarowy wektor obserwowanych cech) do jednej z k klas.
- Tabela krzyżowa (kontyngencji) - tabela przedstawiająca łączny rozkład dwóch lub większej liczby zmiennych. W przykładzie wartości w kolumnie sumują się do 100%, w wierszu już nie muszą.
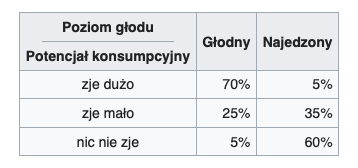
- Estymacja błędu - dzielimy liczbę błędów przez sumę liczby klasyfikacji.
- Szacowanie jakości klasyfikatora:
- Metoda ponownego podstawienia. Korzystając z całej próby (uczącej) uzyskujemy klasyfikator d. Następnie, wykorzystując ten klasyfikator, prognmozujemy etykiety wszystkich elementów próby (obiektów). Miarą jakości jest proporcja poprawnie zaklasyfikowanych obiektów.
- Sprawdzian krzyżowy – metoda statystyczna polegająca na podziale próby statystycznej na podzbiory, a następnie przeprowadzaniu wszelkich analiz na niektórych z nich, tzw. zbiór uczący, podczas gdy pozostałe służą do potwierdzenia wiarygodności jej wyników, tzw. zbiór testowy. [link](https://pl.wikipedia.org/wiki/Sprawdzian_krzyżowy)
- Jak robię lda i dostaję wynik w którym są "coefficients of linear discriminants" to program robi tak że mnoży odpowiednią wartość współczynnika z odpowiednią wartością obserwacji i otrzymany wynik jest używany potem do algorytmu klasyfikacji.
- A posteriori (łac. „z następstwa”) – w filozofii termin będący przeciwieństwem wyrażenia a priori, oznaczający tyle co: "po fakcie".
- Wzór Bayesa

- Bayesowska reguła klasyfikacyjna
