72 KiB
Uczenie maszynowe – zastosowania
10. Sieci neuronowe – propagacja wsteczna
%matplotlib inline
import numpy as np
import math10.1. Metoda propagacji wstecznej – wprowadzenie
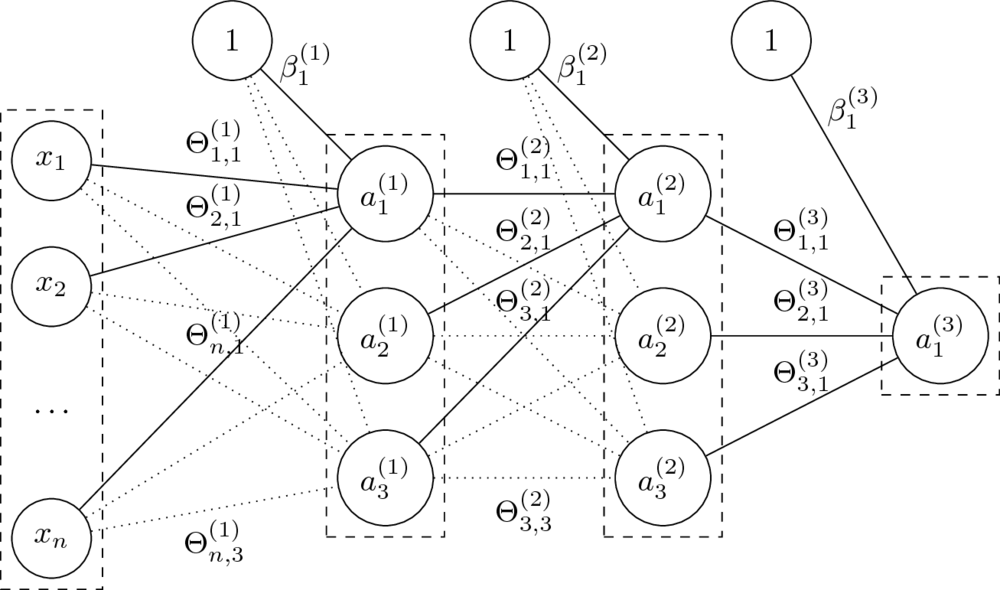
Architektura sieci neuronowych
- Budowa warstwowa, najczęściej sieci jednokierunkowe i gęste.
- Liczbę i rozmiar warstw dobiera się do każdego problemu.
- Rozmiary sieci określane poprzez liczbę neuronów lub parametrów.
_Feedforward
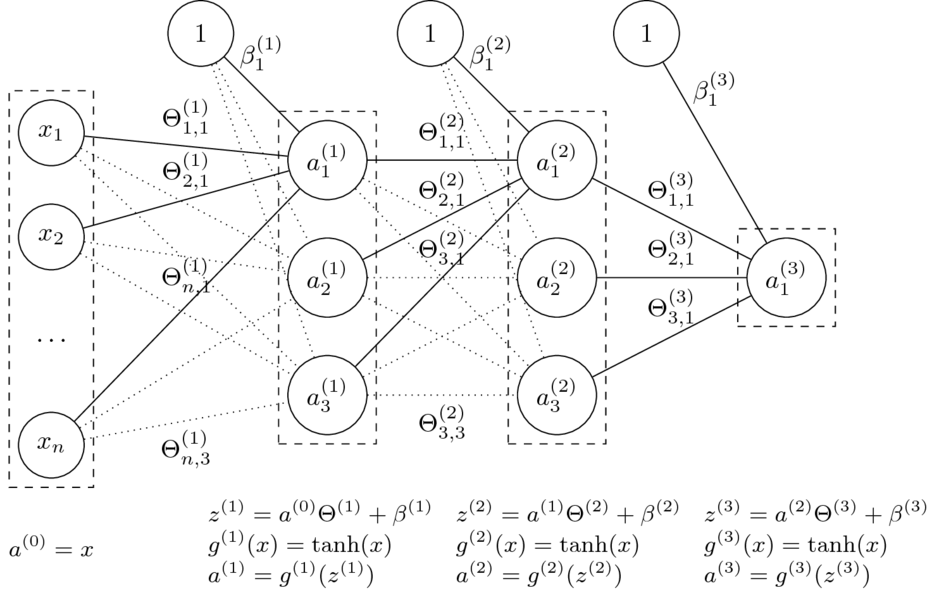
Mając daną $n$-warstwową sieć neuronową oraz jej parametry $\Theta^{(1)}, \ldots, \Theta^{(L)} $ oraz $\beta^{(1)}, \ldots, \beta^{(L)} $, obliczamy:
$$a^{(l)} = g^{(l)}\left( a^{(l-1)} \Theta^{(l)} + \beta^{(l)} \right). $$
- Funkcje $g^{(l)}$ to funkcje aktywacji.
Dla $i = 0$ przyjmujemy $a^{(0)} = x$ (wektor wierszowy cech) oraz $g^{(0)}(x) = x$ (identyczność).
- Parametry $\Theta$ to wagi na połączeniach miedzy neuronami dwóch warstw.
Rozmiar macierzy $\Theta^{(l)}$, czyli macierzy wag na połączeniach warstw $a^{(l-1)}$ i $a^{(l)}$, to $\dim(a^{(l-1)}) \times \dim(a^{(l)})$.
- Parametry $\beta$ zastępują tutaj dodawanie kolumny z jedynkami do macierzy cech.
Macierz $\beta^{(l)}$ ma rozmiar równy liczbie neuronów w odpowiedniej warstwie, czyli $1 \times \dim(a^{(l)})$.
- Klasyfikacja: dla ostatniej warstwy $L$ (o rozmiarze równym liczbie klas) przyjmuje się $g^{(L)}(x) = \mathop{\mathrm{softmax}}(x)$.
- Regresja: pojedynczy neuron wyjściowy; funkcją aktywacji może wtedy być np. funkcja identycznościowa.
- Pozostałe funkcje aktywacji najcześciej mają postać sigmoidy, np. sigmoidalna, tangens hiperboliczny.
Ale niekoniecznie, np. ReLU, leaky ReLU, maxout.
Jak uczyć sieci neuronowe?
- W poznanych do tej pory algorytmach (regresja liniowa, regresja logistyczna) do uczenia używaliśmy funkcji kosztu, jej gradientu oraz algorytmu gradientu prostego (GD/SGD)
- Dla sieci neuronowych potrzebowalibyśmy również znaleźć gradient funkcji kosztu.
- Co sprowadza się do bardziej ogólnego problemu:
jak obliczyć gradient $\nabla f(x)$ dla danej funkcji $f$ i wektora wejściowego $x$?
Pochodna funkcji
- Pochodna mierzy, jak szybko zmienia się wartość funkcji względem zmiany jej argumentów:
$$ \frac{d f(x)}{d x} = \lim_{h \to 0} \frac{ f(x + h) - f(x) }{ h } $$
Pochodna cząstkowa i gradient
- Pochodna cząstkowa mierzy, jak szybko zmienia się wartość funkcji względem zmiany jej _pojedynczego argumentu.
- Gradient to wektor pochodnych cząstkowych:
$$ \nabla f = \left( \frac{\partial f}{\partial x_1}, \ldots, \frac{\partial f}{\partial x_n} \right) $$
Gradient – przykłady
$$ f(x_1, x_2) = x_1 + x_2 \qquad \to \qquad \frac{\partial f}{\partial x_1} = 1, \quad \frac{\partial f}{\partial x_2} = 1, \quad \nabla f = (1, 1) $$
$$ f(x_1, x_2) = x_1 \cdot x_2 \qquad \to \qquad \frac{\partial f}{\partial x_1} = x_2, \quad \frac{\partial f}{\partial x_2} = x_1, \quad \nabla f = (x_2, x_1) $$
$$ f(x_1, x_2) = \max(x_1, x_2) \hskip{12em} \\ \to \qquad \frac{\partial f}{\partial x_1} = \mathbb{1}_{x \geq y}, \quad \frac{\partial f}{\partial x_2} = \mathbb{1}{y \geq x}, \quad \nabla f = (\mathbb{1}{x \geq y}, \mathbb{1}{y \geq x}) $$
Własności pochodnych cząstkowych
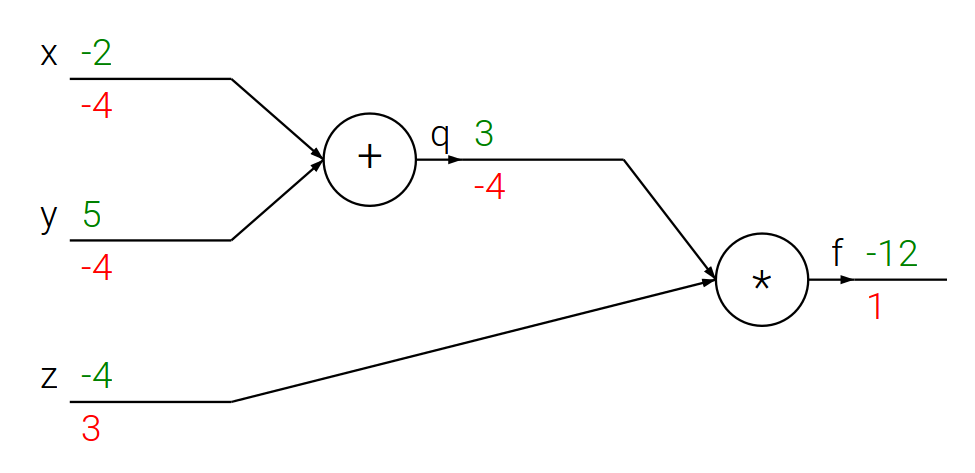
Jezeli $f(x, y, z) = (x + y) , z$ oraz $x + y = q$, to: $$f = q z, \quad \frac{\partial f}{\partial q} = z, \quad \frac{\partial f}{\partial z} = q, \quad \frac{\partial q}{\partial x} = 1, \quad \frac{\partial q}{\partial y} = 1 $$
Reguła łańcuchowa
$$ \frac{\partial f}{\partial x} = \frac{\partial f}{\partial q} , \frac{\partial q}{\partial x}, \quad \frac{\partial f}{\partial y} = \frac{\partial f}{\partial q} , \frac{\partial q}{\partial y} $$
Propagacja wsteczna – prosty przykład
# Dla ustalonego wejścia
x = -2; y = 5; z = -4# Krok w przód
q = x + y
f = q * z
print(q, f)(3, -12)
# UWAGA: teraz za pomocą zmiennych `dfx`, `dfy`, `dfz` i `dfq`
# oznaczę pochodne cząstkowe ∂f/∂x, ∂f/∂y, ∂f/∂z i ∂f/∂q odpowiednio
# Propagacja wsteczna dla f = q * z
dfz = q
dfq = z
# Propagacja wsteczna dla q = x + y
dfx = 1 * dfq # z reguły łańcuchowej
dfy = 1 * dfq # z reguły łańcuchowej
print([dfx, dfy, dfz])[-4, -4, 3]
- Właśnie tak wygląda obliczanie pochodnych metodą propagacji wstecznej!
- Spróbujmy czegoś bardziej skomplikowanego:
metodą propagacji wstecznej obliczmy pochodną funkcji sigmoidalnej.
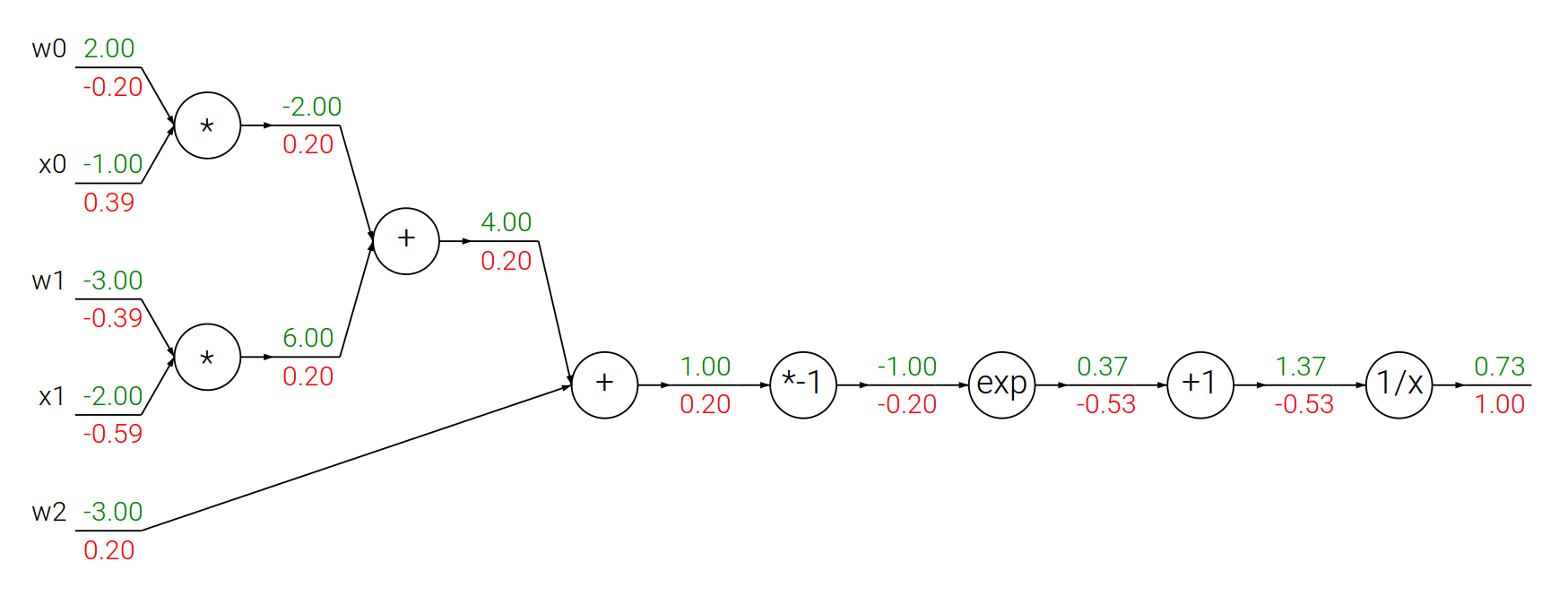
Propagacja wsteczna – funkcja sigmoidalna
Funkcja sigmoidalna:
$$f(\theta,x) = \frac{1}{1+e^{-(\theta_0 x_0 + \theta_1 x_1 + \theta_2)}}$$
$$ \begin{array}{lcl} f(x) = \frac{1}{x} \quad & \rightarrow & \quad \frac{df}{dx} = -\frac{1}{x^2} \\ f_c(x) = c + x \quad & \rightarrow & \quad \frac{df}{dx} = 1 \\ f(x) = e^x \quad & \rightarrow & \quad \frac{df}{dx} = e^x \\ f_a(x) = ax \quad & \rightarrow & \quad \frac{df}{dx} = a \\ \end{array} $$
# Losowe wagi i dane
w = [2,-3,-3]
x = [-1, -2]
# Krok w przód
dot = w[0]*x[0] + w[1]*x[1] + w[2]
f = 1.0 / (1 + math.exp(-dot)) # funkcja sigmoidalna
# Krok w tył
ddot = (1 - f) * f # pochodna funkcji sigmoidalnej
dx = [w[0] * ddot, w[1] * ddot]
dw = [x[0] * ddot, x[1] * ddot, 1.0 * ddot]
print(dx)
print(dw)[0.3932238664829637, -0.5898357997244456] [-0.19661193324148185, -0.3932238664829637, 0.19661193324148185]
Obliczanie gradientów – podsumowanie
- Gradient $f$ dla $x$ mówi jak zmieni się całe wyrażenie przy zmianie wartości $x$.
- Gradienty łączymy korzystając z reguły łańcuchowej.
- W kroku wstecz gradienty informują, które części grafu powinny być zwiększone lub zmniejszone (i z jaką siłą), aby zwiększyć wartość na wyjściu.
- W kontekście implementacji chcemy dzielić funkcję $f$ na części, dla których można łatwo obliczyć gradienty.
10.2. Uczenie wielowarstwowych sieci neuronowych metodą propagacji wstecznej
Mając algorytm SGD oraz gradienty wszystkich wag, moglibyśmy trenować każdą sieć.
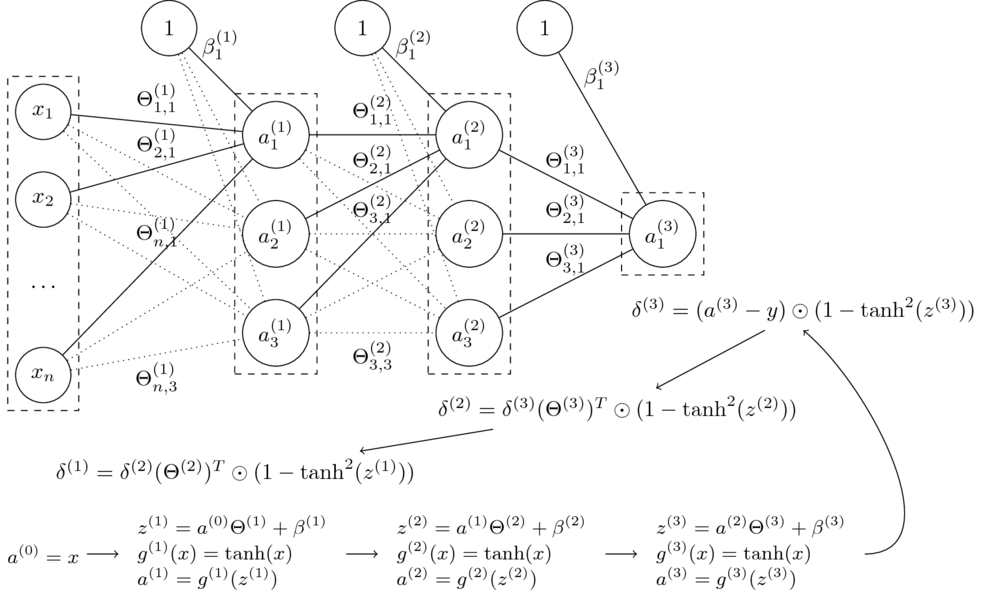
Niech: $$\Theta = (\Theta^{(1)},\Theta^{(2)},\Theta^{(3)},\beta^{(1)},\beta^{(2)},\beta^{(3)})$$
Funkcja sieci neuronowej z grafiki:
$$\small h_\Theta(x) = \tanh(\tanh(\tanh(x\Theta^{(1)}+\beta^{(1)})\Theta^{(2)} + \beta^{(2)})\Theta^{(3)} + \beta^{(3)})$$
- Funkcja kosztu dla regresji: $$J(\Theta) = \dfrac{1}{2m} \sum_{i=1}^{m} (h_\Theta(x^{(i)})- y^{(i)})^2 $$
- Jak obliczymy gradienty?
$$\nabla_{\Theta^{(l)}} J(\Theta) = ? \quad \nabla_{\beta^{(l)}} J(\Theta) = ?$$
W kierunku propagacji wstecznej
- Pewna (niewielka) zmiana wagi $\Delta z^l_j$ dla $j$-ego neuronu w warstwie $l$ pociąga za sobą (niewielką) zmianę kosztu:
$$\frac{\partial J(\Theta)}{\partial z^{l}_j} \Delta z^{l}_j$$
- Jeżeli $\frac{\partial J(\Theta)}{\partial z^{l}_j}$ jest duża, $\Delta z^l_j$ ze znakiem przeciwnym zredukuje koszt.
- Jeżeli $\frac{\partial J(\Theta)}{\partial z^l_j}$ jest bliska zeru, koszt nie będzie mocno poprawiony.
- Definiujemy błąd $\delta^l_j$ neuronu $j$ w warstwie $l$:
$$\delta^l_j := \dfrac{\partial J(\Theta)}{\partial z^l_j}$$ $$\delta^l := \nabla_{z^l} J(\Theta) \quad \textrm{ (zapis wektorowy)} $$
Podstawowe równania propagacji wstecznej
$$ \begin{array}{rcll} \delta^L & = & \nabla_{a^L}J(\Theta) \odot { \left( g^{L} \right) }^{\prime} \left( z^L \right) & (BP1) \\[2mm] \delta^{l} & = & \left( \left( \Theta^{l+1} \right) ! ^\top , \delta^{l+1} \right) \odot {{ \left( g^{l} \right) }^{\prime}} \left( z^{l} \right) & (BP2)\\[2mm] \nabla_{\beta^l} J(\Theta) & = & \delta^l & (BP3)\\[2mm] \nabla_{\Theta^l} J(\Theta) & = & a^{l-1} \odot \delta^l & (BP4)\\ \end{array} $$
(BP1)
$$ \delta^L_j ; = ; \frac{ \partial J }{ \partial a^L_j } , g' !! \left( z^L_j \right) $$ $$ \delta^L ; = ; \nabla_{a^L}J(\Theta) \odot { \left( g^{L} \right) }^{\prime} \left( z^L \right) $$ Błąd w ostatniej warstwie jest iloczynem szybkości zmiany kosztu względem $j$-tego wyjścia i szybkości zmiany funkcji aktywacji w punkcie $z^L_j$.
(BP2)
$$ \delta^{l} ; = ; \left( \left( \Theta^{l+1} \right) ! ^\top , \delta^{l+1} \right) \odot {{ \left( g^{l} \right) }^{\prime}} \left( z^{l} \right) $$ Aby obliczyć błąd w $l$-tej warstwie, należy przemnożyć błąd z następnej ($(l+1)$-szej) warstwy przez transponowany wektor wag, a uzyskaną macierz pomnożyć po współrzędnych przez szybkość zmiany funkcji aktywacji w punkcie $z^l$.
(BP3)
$$ \nabla_{\beta^l} J(\Theta) ; = ; \delta^l $$ Błąd w $l$-tej warstwie jest równy wartości gradientu funkcji kosztu.
(BP4)
$$ \nabla_{\Theta^l} J(\Theta) ; = ; a^{l-1} \odot \delta^l $$ Gradient funkcji kosztu względem wag $l$-tej warstwy można obliczyć jako iloczyn po współrzędnych $a^{l-1}$ przez $\delta^l$.
Algorytm propagacji wstecznej
Dla jednego przykładu $(x,y)$:
- Wejście: Ustaw aktywacje w warstwie cech $a^{(0)}=x$
- Feedforward: dla $l=1,\dots,L$ oblicz $$z^{(l)} = a^{(l-1)} \Theta^{(l)} + \beta^{(l)} \textrm{ oraz } a^{(l)}=g^{(l)} !! \left( z^{(l)} \right) $$
- Błąd wyjścia $\delta^{(L)}$: oblicz wektor $$\delta^{(L)}= \nabla_{a^{(L)}}J(\Theta) \odot {g^{\prime}}^{(L)} !! \left( z^{(L)} \right) $$
- Propagacja wsteczna błędu: dla $l = L-1,L-2,\dots,1$ oblicz $$\delta^{(l)} = \delta^{(l+1)}(\Theta^{(l+1)})^T \odot {g^{\prime}}^{(l)} !! \left( z^{(l)} \right) $$
- Gradienty:
- $\dfrac{\partial}{\partial \Theta_{ij}^{(l)}} J(\Theta) = a_i^{(l-1)}\delta_j^{(l)} \textrm{ oraz } \dfrac{\partial}{\partial \beta_{j}^{(l)}} J(\Theta) = \delta_j^{(l)}$
W naszym przykładzie:
$$\small J(\Theta) = \frac{1}{2} \left( a^{(L)} - y \right) ^2 $$ $$\small \dfrac{\partial}{\partial a^{(L)}} J(\Theta) = a^{(L)} - y$$
$$\small \tanh^{\prime}(x) = 1 - \tanh^2(x)$$
Algorytm SGD z propagacją wsteczną
Pojedyncza iteracja:
- Dla parametrów $\Theta = (\Theta^{(1)},\ldots,\Theta^{(L)})$ utwórz pomocnicze macierze zerowe $\Delta = (\Delta^{(1)},\ldots,\Delta^{(L)})$ o takich samych wymiarach (dla uproszczenia opuszczono wagi $\beta$).
- Dla $m$ przykładów we wsadzie (_batch), $i = 1,\ldots,m$:
- Wykonaj algortym propagacji wstecznej dla przykładu $(x^{(i)}, y^{(i)})$ i przechowaj gradienty $\nabla_{\Theta}J^{(i)}(\Theta)$ dla tego przykładu;
- $\Delta := \Delta + \dfrac{1}{m}\nabla_{\Theta}J^{(i)}(\Theta)$
- Wykonaj aktualizację wag: $\Theta := \Theta - \alpha \Delta$
Propagacja wsteczna – podsumowanie
- Algorytm pierwszy raz wprowadzony w latach 70. XX w.
- W 1986 David Rumelhart, Geoffrey Hinton i Ronald Williams pokazali, że jest znacznie szybszy od wcześniejszych metod.
- Obecnie najpopularniejszy algorytm uczenia sieci neuronowych.
10.3. Przykłady implementacji wielowarstwowych sieci neuronowych
Uwaga!
Poniższe przykłady wykorzystują interfejs Keras, który jest częścią biblioteki TensorFlow.
Aby uruchomić TensorFlow w środowisku Jupyter, należy wykonać następujące czynności:
Przed pierwszym uruchomieniem (wystarczy wykonać tylko raz)
Instalacja biblioteki TensorFlow w środowisku Anaconda:
- Uruchom _Anaconda Navigator
- Wybierz kafelek _CMD.exe Prompt
- Kliknij przycisk _Launch
- Pojawi się konsola. Wpisz następujące polecenia, każde zatwierdzając wciśnięciem klawisza Enter:
conda create -n tf tensorflow conda activate tf conda install pandas matplotlib jupyter notebook
Przed każdym uruchomieniem
Jeżeli chcemy korzystać z biblioteki TensorFlow, to środowisko Jupyter Notebook należy uruchomić w następujący sposób:
- Uruchom _Anaconda Navigator
- Wybierz kafelek _CMD.exe Prompt
- Kliknij przycisk _Launch
- Pojawi się konsola. Wpisz następujące polecenia, każde zatwierdzając wciśnięciem klawisza Enter:
conda activate tf jupyter notebook
Przykład: MNIST
_Modified National Institute of Standards and Technology database
- Zbiór cyfr zapisanych pismem odręcznym
- 60 000 przykładów uczących, 10 000 przykładów testowych
- Rozdzielczość każdego przykładu: 28 × 28 = 784 piksele
# źródło: https://github.com/keras-team/keras/examples/minst_mlp.py
from tensorflow import keras
from tensorflow.keras.datasets import mnist
from tensorflow.keras.layers import Dense, Dropout
# załaduj dane i podziel je na zbiory uczący i testowy
(x_train, y_train), (x_test, y_test) = mnist.load_data()from matplotlib import pyplot as plt
def draw_examples(examples, captions=None):
plt.figure(figsize=(16, 4))
m = len(examples)
for i, example in enumerate(examples):
plt.subplot(100 + m * 10 + i + 1)
plt.imshow(example, cmap=plt.get_cmap('gray'))
plt.show()
if captions is not None:
print(6 * ' ' + (10 * ' ').join(str(captions[i]) for i in range(m)))draw_examples(x_train[:7], captions=y_train)5 0 4 1 9 2 1
num_classes = 10
x_train = x_train.reshape(60000, 784) # 784 = 28 * 28
x_test = x_test.reshape(10000, 784)
x_train = x_train.astype('float32')
x_test = x_test.astype('float32')
x_train /= 255
x_test /= 255
print('{} przykładów uczących'.format(x_train.shape[0]))
print('{} przykładów testowych'.format(x_test.shape[0]))
# przekonwertuj wektory klas na binarne macierze klas
y_train = keras.utils.to_categorical(y_train, num_classes)
y_test = keras.utils.to_categorical(y_test, num_classes)60000 przykładów uczących 10000 przykładów testowych
model = keras.Sequential()
model.add(Dense(512, activation='tanh', input_shape=(784,)))
model.add(Dense(512, activation='tanh'))
model.add(Dense(num_classes, activation='softmax'))
model.summary() # wyświetl podsumowanie architektury sieciModel: "sequential" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense (Dense) (None, 512) 401920 _________________________________________________________________ dense_1 (Dense) (None, 512) 262656 _________________________________________________________________ dense_2 (Dense) (None, 10) 5130 ================================================================= Total params: 669,706 Trainable params: 669,706 Non-trainable params: 0 _________________________________________________________________
print(x_train.shape, y_train.shape)(60000, 784) (60000, 10)
print(y_train[:10])[[0. 0. 0. 0. 0. 1. 0. 0. 0. 0.] [1. 0. 0. 0. 0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 1. 0. 0. 0. 0. 0.] [0. 1. 0. 0. 0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 0. 0. 0. 0. 0. 1.] [0. 0. 1. 0. 0. 0. 0. 0. 0. 0.] [0. 1. 0. 0. 0. 0. 0. 0. 0. 0.] [0. 0. 0. 1. 0. 0. 0. 0. 0. 0.] [0. 1. 0. 0. 0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 1. 0. 0. 0. 0. 0.]]
model.compile(loss='categorical_crossentropy', optimizer=keras.optimizers.Adam(), metrics=['accuracy'])
model.fit(x_train, y_train, batch_size=128, epochs=10, verbose=1,
validation_data=(x_test, y_test))Epoch 1/10 469/469 [==============================] - 11s 24ms/step - loss: 0.2807 - accuracy: 0.9158 - val_loss: 0.1509 - val_accuracy: 0.9550 Epoch 2/10 469/469 [==============================] - 11s 24ms/step - loss: 0.1242 - accuracy: 0.9619 - val_loss: 0.1076 - val_accuracy: 0.9677 Epoch 3/10 469/469 [==============================] - 11s 24ms/step - loss: 0.0812 - accuracy: 0.9752 - val_loss: 0.0862 - val_accuracy: 0.9723 Epoch 4/10 469/469 [==============================] - 11s 24ms/step - loss: 0.0587 - accuracy: 0.9820 - val_loss: 0.0823 - val_accuracy: 0.9727 Epoch 5/10 469/469 [==============================] - 11s 24ms/step - loss: 0.0416 - accuracy: 0.9870 - val_loss: 0.0735 - val_accuracy: 0.9763 Epoch 6/10 469/469 [==============================] - 11s 24ms/step - loss: 0.0318 - accuracy: 0.9897 - val_loss: 0.0723 - val_accuracy: 0.9761s: 0.0318 - accuracy: Epoch 7/10 469/469 [==============================] - 11s 23ms/step - loss: 0.0215 - accuracy: 0.9940 - val_loss: 0.0685 - val_accuracy: 0.9792 Epoch 8/10 469/469 [==============================] - 11s 23ms/step - loss: 0.0189 - accuracy: 0.9943 - val_loss: 0.0705 - val_accuracy: 0.9786 Epoch 9/10 469/469 [==============================] - 11s 24ms/step - loss: 0.0148 - accuracy: 0.9957 - val_loss: 0.0674 - val_accuracy: 0.9790 Epoch 10/10 469/469 [==============================] - 11s 23ms/step - loss: 0.0092 - accuracy: 0.9978 - val_loss: 0.0706 - val_accuracy: 0.9798
<tensorflow.python.keras.callbacks.History at 0x1bde5f96b50>
score = model.evaluate(x_test, y_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.07055816799402237 Test accuracy: 0.9797999858856201
Warstwa _dropout to metoda regularyzacji, służy zapobieganiu nadmiernemu dopasowaniu sieci. Polega na tym, że część węzłów sieci jest usuwana w sposób losowy.
# Bez warstw Dropout
num_classes = 10
(x_train, y_train), (x_test, y_test) = mnist.load_data()
x_train = x_train.reshape(60000, 784) # 784 = 28 * 28
x_test = x_test.reshape(10000, 784)
x_train = x_train.astype('float32')
x_test = x_test.astype('float32')
x_train /= 255
x_test /= 255
y_train = keras.utils.to_categorical(y_train, num_classes)
y_test = keras.utils.to_categorical(y_test, num_classes)
model_no_dropout = keras.Sequential()
model_no_dropout.add(Dense(512, activation='relu', input_shape=(784,)))
model_no_dropout.add(Dense(512, activation='relu'))
model_no_dropout.add(Dense(num_classes, activation='softmax'))
model_no_dropout.summary()
model_no_dropout.compile(loss='categorical_crossentropy',
optimizer=keras.optimizers.RMSprop(),
metrics=['accuracy'])
model_no_dropout.fit(x_train, y_train,
batch_size=128,
epochs=5,
verbose=1,
validation_data=(x_test, y_test))Model: "sequential_22" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_62 (Dense) (None, 512) 401920 _________________________________________________________________ dense_63 (Dense) (None, 512) 262656 _________________________________________________________________ dense_64 (Dense) (None, 10) 5130 ================================================================= Total params: 669,706 Trainable params: 669,706 Non-trainable params: 0 _________________________________________________________________ Epoch 1/5 469/469 [==============================] - 10s 20ms/step - loss: 0.2203 - accuracy: 0.9317 - val_loss: 0.0936 - val_accuracy: 0.9697 Epoch 2/5 469/469 [==============================] - 10s 21ms/step - loss: 0.0816 - accuracy: 0.9746 - val_loss: 0.0747 - val_accuracy: 0.9779 Epoch 3/5 469/469 [==============================] - 10s 20ms/step - loss: 0.0544 - accuracy: 0.9827 - val_loss: 0.0674 - val_accuracy: 0.9798 Epoch 4/5 469/469 [==============================] - 10s 22ms/step - loss: 0.0384 - accuracy: 0.9879 - val_loss: 0.0746 - val_accuracy: 0.9806 Epoch 5/5 469/469 [==============================] - 10s 22ms/step - loss: 0.0298 - accuracy: 0.9901 - val_loss: 0.0736 - val_accuracy: 0.9801
<tensorflow.python.keras.callbacks.History at 0x1ed7eba8070>
# Bez warstw Dropout
score = model_no_dropout.evaluate(x_test, y_test, verbose=0)
print('Test loss (no dropout): {}'.format(score[0]))
print('Test accuracy (no dropout): {}'.format(score[1]))Test loss (no dropout): 0.07358124107122421 Test accuracy (no dropout): 0.9800999760627747
# Więcej warstw, inna funkcja aktywacji
num_classes = 10
(x_train, y_train), (x_test, y_test) = mnist.load_data()
x_train = x_train.reshape(60000, 784) # 784 = 28 * 28
x_test = x_test.reshape(10000, 784)
x_train = x_train.astype('float32')
x_test = x_test.astype('float32')
x_train /= 255
x_test /= 255
y_train = keras.utils.to_categorical(y_train, num_classes)
y_test = keras.utils.to_categorical(y_test, num_classes)
model3 = Sequential()
model3.add(Dense(2500, activation='tanh', input_shape=(784,)))
model3.add(Dense(2000, activation='tanh'))
model3.add(Dense(1500, activation='tanh'))
model3.add(Dense(1000, activation='tanh'))
model3.add(Dense(500, activation='tanh'))
model3.add(Dense(num_classes, activation='softmax'))
model3.summary()
model3.compile(loss='categorical_crossentropy',
optimizer=keras.optimizers.RMSprop(),
metrics=['accuracy'])
model3.fit(x_train, y_train,
batch_size=128,
epochs=10,
verbose=1,
validation_data=(x_test, y_test))Model: "sequential_23" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_65 (Dense) (None, 2500) 1962500 _________________________________________________________________ dense_66 (Dense) (None, 2000) 5002000 _________________________________________________________________ dense_67 (Dense) (None, 1500) 3001500 _________________________________________________________________ dense_68 (Dense) (None, 1000) 1501000 _________________________________________________________________ dense_69 (Dense) (None, 500) 500500 _________________________________________________________________ dense_70 (Dense) (None, 10) 5010 ================================================================= Total params: 11,972,510 Trainable params: 11,972,510 Non-trainable params: 0 _________________________________________________________________ Epoch 1/10 469/469 [==============================] - 129s 275ms/step - loss: 0.9587 - accuracy: 0.7005 - val_loss: 0.5066 - val_accuracy: 0.8566 Epoch 2/10 469/469 [==============================] - 130s 276ms/step - loss: 0.2666 - accuracy: 0.9234 - val_loss: 0.3376 - val_accuracy: 0.9024 Epoch 3/10 469/469 [==============================] - 130s 277ms/step - loss: 0.1811 - accuracy: 0.9477 - val_loss: 0.1678 - val_accuracy: 0.9520 Epoch 4/10 469/469 [==============================] - 134s 287ms/step - loss: 0.1402 - accuracy: 0.9588 - val_loss: 0.1553 - val_accuracy: 0.9576 Epoch 5/10 469/469 [==============================] - 130s 278ms/step - loss: 0.1153 - accuracy: 0.9662 - val_loss: 0.1399 - val_accuracy: 0.9599 Epoch 6/10 469/469 [==============================] - 130s 277ms/step - loss: 0.0956 - accuracy: 0.9711 - val_loss: 0.1389 - val_accuracy: 0.9612 Epoch 7/10 469/469 [==============================] - 131s 280ms/step - loss: 0.0803 - accuracy: 0.9761 - val_loss: 0.1008 - val_accuracy: 0.9724 Epoch 8/10 469/469 [==============================] - 134s 286ms/step - loss: 0.0685 - accuracy: 0.9797 - val_loss: 0.1137 - val_accuracy: 0.9679 Epoch 9/10 469/469 [==============================] - 130s 278ms/step - loss: 0.0602 - accuracy: 0.9819 - val_loss: 0.1064 - val_accuracy: 0.9700 Epoch 10/10 469/469 [==============================] - 129s 274ms/step - loss: 0.0520 - accuracy: 0.9843 - val_loss: 0.1095 - val_accuracy: 0.9698
<tensorflow.python.keras.callbacks.History at 0x1ed0e628250>
# Więcej warstw, inna funkcja aktywacji
score = model3.evaluate(x_test, y_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.10945799201726913 Test accuracy: 0.9697999954223633
Przykład: 4-pikselowy aparat fotograficzny
def generate_example(description):
variant = random.choice([1, -1])
if description == 's': # solid
return (np.array([[ 1.0, 1.0], [ 1.0, 1.0]]) if variant == 1 else
np.array([[-1.0, -1.0], [-1.0, -1.0]]))
elif description == 'v': # vertical
return (np.array([[ 1.0, -1.0], [ 1.0, -1.0]]) if variant == 1 else
np.array([[-1.0, 1.0], [-1.0, 1.0]]))
elif description == 'd': # diagonal
return (np.array([[ 1.0, -1.0], [-1.0, 1.0]]) if variant == 1 else
np.array([[-1.0, 1.0], [ 1.0, -1.0]]))
elif description == 'h': # horizontal
return (np.array([[ 1.0, 1.0], [-1.0, -1.0]]) if variant == 1 else
np.array([[-1.0, -1.0], [ 1.0, 1.0]]))
else:
return np.array([[random.uniform(-1, 1), random.uniform(-1, 1)],
[random.uniform(-1, 1), random.uniform(-1, 1)]])import random
num_classes = 4
trainset_size = 4000
testset_size = 1000
y4_train = np.array([random.choice(['s', 'v', 'd', 'h']) for i in range(trainset_size)])
x4_train = np.array([generate_example(desc) for desc in y4_train])
y4_test = np.array([random.choice(['s', 'v', 'd', 'h']) for i in range(testset_size)])
x4_test = np.array([generate_example(desc) for desc in y4_test])draw_examples(x4_train[:7], captions=y4_train)s d h s d v v
x4_train = x4_train.reshape(trainset_size, 4)
x4_test = x4_test.reshape(testset_size, 4)
x4_train = x4_train.astype('float32')
x4_test = x4_test.astype('float32')
y4_train = np.array([{'s': 0, 'v': 1, 'd': 2, 'h': 3}[desc] for desc in y4_train])
y4_test = np.array([{'s': 0, 'v': 1, 'd': 2, 'h': 3}[desc] for desc in y4_test])
y4_train = keras.utils.to_categorical(y4_train, num_classes)
y4_test = keras.utils.to_categorical(y4_test, num_classes)model4 = keras.Sequential()
model4.add(Dense(4, activation='tanh', input_shape=(4,)))
model4.add(Dense(4, activation='tanh'))
model4.add(Dense(8, activation='relu'))
model4.add(Dense(num_classes, activation='softmax'))
model4.summary()Model: "sequential_24" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_71 (Dense) (None, 4) 20 _________________________________________________________________ dense_72 (Dense) (None, 4) 20 _________________________________________________________________ dense_73 (Dense) (None, 8) 40 _________________________________________________________________ dense_74 (Dense) (None, 4) 36 ================================================================= Total params: 116 Trainable params: 116 Non-trainable params: 0 _________________________________________________________________
model4.layers[0].set_weights(
[np.array([[ 1.0, 0.0, 1.0, 0.0],
[ 0.0, 1.0, 0.0, 1.0],
[ 1.0, 0.0, -1.0, 0.0],
[ 0.0, 1.0, 0.0, -1.0]],
dtype=np.float32), np.array([0., 0., 0., 0.], dtype=np.float32)])
model4.layers[1].set_weights(
[np.array([[ 1.0, -1.0, 0.0, 0.0],
[ 1.0, 1.0, 0.0, 0.0],
[ 0.0, 0.0, 1.0, -1.0],
[ 0.0, 0.0, -1.0, -1.0]],
dtype=np.float32), np.array([0., 0., 0., 0.], dtype=np.float32)])
model4.layers[2].set_weights(
[np.array([[ 1.0, -1.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0],
[ 0.0, 0.0, 1.0, -1.0, 0.0, 0.0, 0.0, 0.0],
[ 0.0, 0.0, 0.0, 0.0, 1.0, -1.0, 0.0, 0.0],
[ 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 1.0, -1.0]],
dtype=np.float32), np.array([0., 0., 0., 0., 0., 0., 0., 0.], dtype=np.float32)])model4.layers[3].set_weights(
[np.array([[ 1.0, 0.0, 0.0, 0.0],
[ 1.0, 0.0, 0.0, 0.0],
[ 0.0, 1.0, 0.0, 0.0],
[ 0.0, 1.0, 0.0, 0.0],
[ 0.0, 0.0, 1.0, 0.0],
[ 0.0, 0.0, 1.0, 0.0],
[ 0.0, 0.0, 0.0, 1.0],
[ 0.0, 0.0, 0.0, 1.0]],
dtype=np.float32), np.array([0., 0., 0., 0.], dtype=np.float32)])
model4.compile(loss='categorical_crossentropy',
optimizer=keras.optimizers.Adagrad(),
metrics=['accuracy'])for layer in model4.layers:
print(layer.get_weights())[array([[ 1., 0., 1., 0.],
[ 0., 1., 0., 1.],
[ 1., 0., -1., 0.],
[ 0., 1., 0., -1.]], dtype=float32), array([0., 0., 0., 0.], dtype=float32)]
[array([[ 1., -1., 0., 0.],
[ 1., 1., 0., 0.],
[ 0., 0., 1., -1.],
[ 0., 0., -1., -1.]], dtype=float32), array([0., 0., 0., 0.], dtype=float32)]
[array([[ 1., -1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., -1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., -1., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., -1.]], dtype=float32), array([0., 0., 0., 0., 0., 0., 0., 0.], dtype=float32)]
[array([[1., 0., 0., 0.],
[1., 0., 0., 0.],
[0., 1., 0., 0.],
[0., 1., 0., 0.],
[0., 0., 1., 0.],
[0., 0., 1., 0.],
[0., 0., 0., 1.],
[0., 0., 0., 1.]], dtype=float32), array([0., 0., 0., 0.], dtype=float32)]
model4.predict([np.array([[1.0, 1.0], [-1.0, -1.0]]).reshape(1, 4)])array([[0.17831734, 0.17831734, 0.17831734, 0.465048 ]], dtype=float32)
score = model4.evaluate(x4_test, y4_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.7656148672103882 Test accuracy: 1.0
model5 = Sequential()
model5.add(Dense(4, activation='tanh', input_shape=(4,)))
model5.add(Dense(4, activation='tanh'))
model5.add(Dense(8, activation='relu'))
model5.add(Dense(num_classes, activation='softmax'))
model5.compile(loss='categorical_crossentropy',
optimizer=keras.optimizers.RMSprop(),
metrics=['accuracy'])
model5.summary()Model: "sequential_25" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_75 (Dense) (None, 4) 20 _________________________________________________________________ dense_76 (Dense) (None, 4) 20 _________________________________________________________________ dense_77 (Dense) (None, 8) 40 _________________________________________________________________ dense_78 (Dense) (None, 4) 36 ================================================================= Total params: 116 Trainable params: 116 Non-trainable params: 0 _________________________________________________________________
model5.fit(x4_train, y4_train, epochs=8, validation_data=(x4_test, y4_test))Epoch 1/8 125/125 [==============================] - 0s 3ms/step - loss: 1.3126 - accuracy: 0.3840 - val_loss: 1.1926 - val_accuracy: 0.6110 Epoch 2/8 125/125 [==============================] - 0s 2ms/step - loss: 1.0978 - accuracy: 0.5980 - val_loss: 1.0085 - val_accuracy: 0.6150 Epoch 3/8 125/125 [==============================] - 0s 2ms/step - loss: 0.9243 - accuracy: 0.7035 - val_loss: 0.8416 - val_accuracy: 0.7380 Epoch 4/8 125/125 [==============================] - 0s 2ms/step - loss: 0.7522 - accuracy: 0.8740 - val_loss: 0.6738 - val_accuracy: 1.0000 Epoch 5/8 125/125 [==============================] - 0s 2ms/step - loss: 0.5811 - accuracy: 1.0000 - val_loss: 0.5030 - val_accuracy: 1.0000 Epoch 6/8 125/125 [==============================] - 0s 2ms/step - loss: 0.4134 - accuracy: 1.0000 - val_loss: 0.3428 - val_accuracy: 1.0000 Epoch 7/8 125/125 [==============================] - 0s 2ms/step - loss: 0.2713 - accuracy: 1.0000 - val_loss: 0.2161 - val_accuracy: 1.0000 Epoch 8/8 125/125 [==============================] - 0s 1ms/step - loss: 0.1621 - accuracy: 1.0000 - val_loss: 0.1225 - val_accuracy: 1.0000
<tensorflow.python.keras.callbacks.History at 0x1ed00809700>
model5.predict([np.array([[1.0, 1.0], [-1.0, -1.0]]).reshape(1, 4)])array([[3.2040708e-02, 1.0065207e-03, 4.9596769e-04, 9.6645677e-01]],
dtype=float32)score = model5.evaluate(x4_test, y4_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.1224619448184967 Test accuracy: 1.0
import contextlib
@contextlib.contextmanager
def printoptions(*args, **kwargs):
original = np.get_printoptions()
np.set_printoptions(*args, **kwargs)
try:
yield
finally:
np.set_printoptions(**original)with printoptions(precision=1, suppress=True):
for layer in model5.layers:
print(layer.get_weights())[array([[ 0.7, 0.2, -0.7, 0.7],
[-0.5, 0.9, 0.6, 0.6],
[ 1.1, 0.2, 0.1, 0.2],
[ 0.7, 0.1, 0.3, -0.7]], dtype=float32), array([ 0. , 0.1, -0.1, -0.2], dtype=float32)]
[array([[ 0.7, 0.5, -1.1, -1.2],
[ 0.7, 0.9, -0.6, 0.3],
[ 0.1, 1.4, -0.6, 0.8],
[ 1.5, 0.1, -0.1, 0.9]], dtype=float32), array([-0.4, 0.2, -0. , 0.2], dtype=float32)]
[array([[-1. , 1. , -0.7, -0.3, 0.2, 1.3, -0.7, 0.9],
[-0.9, 0.5, 0.8, -1.3, -1.2, 1.3, 0.4, -1. ],
[ 0.9, 0.2, 0.3, 0.4, 1.3, -0.9, -0.1, -0.2],
[-0.4, 0.5, 1.1, -0.6, 1.1, 0.1, -1.5, -1. ]], dtype=float32), array([-0.1, 0.1, 0.1, 0.1, 0.2, -0. , 0.1, 0.2], dtype=float32)]
[array([[ 0.7, -0.5, 0.8, -0.5],
[-0.3, -1.6, -0.2, 0.1],
[-1.5, 0.9, 0.1, -0.5],
[ 0.6, 0.7, 1. , -1.4],
[ 0.7, -1.2, -1.6, 1.2],
[ 1. , -1.2, 0.3, -1.5],
[-0.2, 0. , 0.6, 1.3],
[-0.8, 0.2, -0.6, -1. ]], dtype=float32), array([-0.6, 0.5, -0.3, 0.4], dtype=float32)]