145 KiB
145 KiB
Uczenie maszynowe UMZ 2019/2020
26 maja 2020
11. Wielowarstwowe sieci neuronowe i algorytmy optymalizacji
11.1. Funkcje aktywacji
- Każda funkcja aktywacji ma swoje zalety i wady.
- Różne rodzaje funkcji aktywacji nadają się do różnych zastosowań.
%matplotlib inline
import math
import matplotlib.pyplot as plt
import numpy as np
import random
import keras
from keras.datasets import mnist
from keras.models import Sequential
from keras.layers import Dense, Dropout, SimpleRNN, LSTM
from keras.optimizers import Adagrad, Adam, RMSprop, SGD
from IPython.display import YouTubeVideo/home/pawel/.local/lib/python2.7/site-packages/h5py/__init__.py:36: FutureWarning: Conversion of the second argument of issubdtype from `float` to `np.floating` is deprecated. In future, it will be treated as `np.float64 == np.dtype(float).type`. from ._conv import register_converters as _register_converters Using TensorFlow backend.
def plot(fun):
x = np.arange(-3.0, 3.0, 0.01)
y = [fun(x_i) for x_i in x]
fig = plt.figure(figsize=(14, 7))
ax = fig.add_subplot(111)
fig.subplots_adjust(left=0.1, right=0.9, bottom=0.1, top=0.9)
ax.set_xlim(-3.0, 3.0)
ax.set_ylim(-1.5, 1.5)
ax.grid()
ax.plot(x, y)
plt.show()Funkcja logistyczna
$$ g(x) = \frac{1}{1 + e^{-x}} $$
- Przyjmuje wartości z przedziału $(0, 1)$.
Funkcja logistyczna – wykres
plot(lambda x: 1 / (1 + math.exp(-x)))Tangens hiperboliczny
$$ g(x) = \tanh x = \frac{e^{x} - e^{-x}}{e^{x} + e^{-x}} $$
- Przyjmuje wartości z przedziału $(-1, 1)$.
- Powstaje z funkcji logistycznej przez przeskalowanie i przesunięcie.
Tangens hiperboliczny – wykres
plot(lambda x: math.tanh(x))ReLU (_Rectifier Linear Unit)
$$ g(x) = \max(0, x) $$
ReLU – zalety
- Mniej podatna na problem zanikającego gradientu (_vanishing gradient) niż funkcje sigmoidalne, dzięki czemu SGD jest szybciej zbieżna.
- Prostsze obliczanie gradientu.
- Dzięki zerowaniu ujemnych wartości, wygasza neurony, „rozrzedzając” sieć (_sparsity), co przyspiesza obliczenia.
ReLU – wady
- Dla dużych wartości gradient może „eksplodować”.
- „Wygaszanie” neuronów.
ReLU – wykres
plot(lambda x: max(0, x))Softplus
$$ g(x) = \log(1 + e^{x}) $$
- Wygładzona wersja ReLU.
Softplus – wykres
plot(lambda x: math.log(1 + math.exp(x)))Problem zanikającego gradientu (_vanishing gradient problem)
- Sigmoidalne funkcje aktywacji ograniczają wartości na wyjściach neuronów do niewielkich przedziałów ($(-1, 1)$, $(0, 1)$ itp.).
- Jeżeli sieć ma wiele warstw, to podczas propagacji wstecznej mnożymy przez siebie wiele małych wartości → obliczony gradient jest mały.
- Im więcej warstw, tym silniejszy efekt zanikania.
Sposoby na zanikający gradient
- Modyfikacja algorytmu optymalizacji (_RProp, RMSProp)
- Użycie innej funckji aktywacji (ReLU, softplus)
- Dodanie warstw _dropout
- Nowe architektury (LSTM itp.)
- Więcej danych, zwiększenie mocy obliczeniowej
11.2. Wielowarstwowe sieci neuronowe w praktyce
Przykład: MNIST
_Modified National Institute of Standards and Technology database
- Zbiór cyfr zapisanych pismem odręcznym
- 60 000 przykładów uczących, 10 000 przykładów testowych
- Rozdzielczość każdego przykładu: 28 × 28 = 784 piksele
# źródło: https://github.com/keras-team/keras/examples/minst_mlp.py
import keras
from keras.datasets import mnist
# załaduj dane i podziel je na zbiory uczący i testowy
(x_train, y_train), (x_test, y_test) = mnist.load_data()def draw_examples(examples, captions=None):
plt.figure(figsize=(16, 4))
m = len(examples)
for i, example in enumerate(examples):
plt.subplot(100 + m * 10 + i + 1)
plt.imshow(example, cmap=plt.get_cmap('gray'))
plt.show()
if captions is not None:
print(6 * ' ' + (10 * ' ').join(str(captions[i]) for i in range(m)))draw_examples(x_train[:7], captions=y_train)5 0 4 1 9 2 1
num_classes = 10
x_train = x_train.reshape(60000, 784) # 784 = 28 * 28
x_test = x_test.reshape(10000, 784)
x_train = x_train.astype('float32')
x_test = x_test.astype('float32')
x_train /= 255
x_test /= 255
print('{} przykładów uczących'.format(x_train.shape[0]))
print('{} przykładów testowych'.format(x_test.shape[0]))
# przekonwertuj wektory klas na binarne macierze klas
y_train = keras.utils.to_categorical(y_train, num_classes)
y_test = keras.utils.to_categorical(y_test, num_classes)60000 przykładów uczących 10000 przykładów testowych
model = Sequential()
model.add(Dense(512, activation='relu', input_shape=(784,)))
model.add(Dropout(0.2))
model.add(Dense(512, activation='relu'))
model.add(Dropout(0.2))
model.add(Dense(num_classes, activation='softmax'))
model.summary()_________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_1 (Dense) (None, 512) 401920 _________________________________________________________________ dropout_1 (Dropout) (None, 512) 0 _________________________________________________________________ dense_2 (Dense) (None, 512) 262656 _________________________________________________________________ dropout_2 (Dropout) (None, 512) 0 _________________________________________________________________ dense_3 (Dense) (None, 10) 5130 ================================================================= Total params: 669,706 Trainable params: 669,706 Non-trainable params: 0 _________________________________________________________________
print(x_train.shape, y_train.shape)((60000, 784), (60000, 10))
model.compile(loss='categorical_crossentropy', optimizer=RMSprop(), metrics=['accuracy'])
model.fit(x_train, y_train, batch_size=128, epochs=5, verbose=1,
validation_data=(x_test, y_test))Train on 60000 samples, validate on 10000 samples Epoch 1/5 60000/60000 [==============================] - 9s 153us/step - loss: 0.2489 - acc: 0.9224 - val_loss: 0.1005 - val_acc: 0.9706 Epoch 2/5 60000/60000 [==============================] - 9s 151us/step - loss: 0.1042 - acc: 0.9683 - val_loss: 0.0861 - val_acc: 0.9740 Epoch 3/5 60000/60000 [==============================] - 9s 153us/step - loss: 0.0742 - acc: 0.9782 - val_loss: 0.0733 - val_acc: 0.9796 Epoch 4/5 60000/60000 [==============================] - 9s 154us/step - loss: 0.0603 - acc: 0.9824 - val_loss: 0.0713 - val_acc: 0.9800 Epoch 5/5 60000/60000 [==============================] - 9s 157us/step - loss: 0.0512 - acc: 0.9848 - val_loss: 0.0749 - val_acc: 0.9795
<keras.callbacks.History at 0x7fdda4f97110>
score = model.evaluate(x_test, y_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.074858742202 Test accuracy: 0.9795
Warstwa _dropout to metoda regularyzacji, służy zapobieganiu nadmiernemu dopasowaniu sieci. Polega na tym, że część węzłów sieci jest usuwana w sposób losowy.
# Bez warstw Dropout
num_classes = 10
(x_train, y_train), (x_test, y_test) = mnist.load_data()
x_train = x_train.reshape(60000, 784) # 784 = 28 * 28
x_test = x_test.reshape(10000, 784)
x_train = x_train.astype('float32')
x_test = x_test.astype('float32')
x_train /= 255
x_test /= 255
y_train = keras.utils.to_categorical(y_train, num_classes)
y_test = keras.utils.to_categorical(y_test, num_classes)
model_no_dropout = Sequential()
model_no_dropout.add(Dense(512, activation='relu', input_shape=(784,)))
model_no_dropout.add(Dense(512, activation='relu'))
model_no_dropout.add(Dense(num_classes, activation='softmax'))
model_no_dropout.summary()
model_no_dropout.compile(loss='categorical_crossentropy',
optimizer=RMSprop(),
metrics=['accuracy'])
model_no_dropout.fit(x_train, y_train,
batch_size=128,
epochs=5,
verbose=1,
validation_data=(x_test, y_test))_________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_4 (Dense) (None, 512) 401920 _________________________________________________________________ dense_5 (Dense) (None, 512) 262656 _________________________________________________________________ dense_6 (Dense) (None, 10) 5130 ================================================================= Total params: 669,706 Trainable params: 669,706 Non-trainable params: 0 _________________________________________________________________ Train on 60000 samples, validate on 10000 samples Epoch 1/5 60000/60000 [==============================] - 8s 139us/step - loss: 0.2237 - acc: 0.9303 - val_loss: 0.0998 - val_acc: 0.9676 Epoch 2/5 60000/60000 [==============================] - 8s 136us/step - loss: 0.0818 - acc: 0.9748 - val_loss: 0.0788 - val_acc: 0.9770 Epoch 3/5 60000/60000 [==============================] - 8s 136us/step - loss: 0.0538 - acc: 0.9831 - val_loss: 0.1074 - val_acc: 0.9695 Epoch 4/5 60000/60000 [==============================] - 10s 161us/step - loss: 0.0397 - acc: 0.9879 - val_loss: 0.0871 - val_acc: 0.9763 Epoch 5/5 60000/60000 [==============================] - 12s 195us/step - loss: 0.0299 - acc: 0.9910 - val_loss: 0.0753 - val_acc: 0.9812
<keras.callbacks.History at 0x7fdda3dcad50>
# Bez warstw Dropout
score = model_no_dropout.evaluate(x_test, y_test, verbose=0)
print('Test loss (no dropout): {}'.format(score[0]))
print('Test accuracy (no dropout): {}'.format(score[1]))Test loss (no dropout): 0.0753162465898 Test accuracy (no dropout): 0.9812
# Więcej warstw, inna funkcja aktywacji
num_classes = 10
(x_train, y_train), (x_test, y_test) = mnist.load_data()
x_train = x_train.reshape(60000, 784) # 784 = 28 * 28
x_test = x_test.reshape(10000, 784)
x_train = x_train.astype('float32')
x_test = x_test.astype('float32')
x_train /= 255
x_test /= 255
y_train = keras.utils.to_categorical(y_train, num_classes)
y_test = keras.utils.to_categorical(y_test, num_classes)
model3 = Sequential()
model3.add(Dense(2500, activation='tanh', input_shape=(784,)))
model3.add(Dense(2000, activation='tanh'))
model3.add(Dense(1500, activation='tanh'))
model3.add(Dense(1000, activation='tanh'))
model3.add(Dense(500, activation='tanh'))
model3.add(Dense(num_classes, activation='softmax'))
model3.summary()
model3.compile(loss='categorical_crossentropy',
optimizer=RMSprop(),
metrics=['accuracy'])
model3.fit(x_train, y_train,
batch_size=128,
epochs=10,
verbose=1,
validation_data=(x_test, y_test))_________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_7 (Dense) (None, 2500) 1962500 _________________________________________________________________ dense_8 (Dense) (None, 2000) 5002000 _________________________________________________________________ dense_9 (Dense) (None, 1500) 3001500 _________________________________________________________________ dense_10 (Dense) (None, 1000) 1501000 _________________________________________________________________ dense_11 (Dense) (None, 500) 500500 _________________________________________________________________ dense_12 (Dense) (None, 10) 5010 ================================================================= Total params: 11,972,510 Trainable params: 11,972,510 Non-trainable params: 0 _________________________________________________________________ Train on 60000 samples, validate on 10000 samples Epoch 1/10 60000/60000 [==============================] - 145s 2ms/step - loss: 1.4242 - acc: 0.5348 - val_loss: 0.4426 - val_acc: 0.8638 Epoch 2/10 60000/60000 [==============================] - 140s 2ms/step - loss: 0.3245 - acc: 0.9074 - val_loss: 0.2231 - val_acc: 0.9360 Epoch 3/10 60000/60000 [==============================] - 137s 2ms/step - loss: 0.1993 - acc: 0.9420 - val_loss: 0.1694 - val_acc: 0.9485 Epoch 4/10 60000/60000 [==============================] - 136s 2ms/step - loss: 0.1471 - acc: 0.9571 - val_loss: 0.1986 - val_acc: 0.9381 Epoch 5/10 60000/60000 [==============================] - 132s 2ms/step - loss: 0.1189 - acc: 0.9650 - val_loss: 0.1208 - val_acc: 0.9658 Epoch 6/10 60000/60000 [==============================] - 131s 2ms/step - loss: 0.0983 - acc: 0.9711 - val_loss: 0.1260 - val_acc: 0.9637 Epoch 7/10 60000/60000 [==============================] - 129s 2ms/step - loss: 0.0818 - acc: 0.9753 - val_loss: 0.0984 - val_acc: 0.9727 Epoch 8/10 60000/60000 [==============================] - 129s 2ms/step - loss: 0.0710 - acc: 0.9784 - val_loss: 0.1406 - val_acc: 0.9597 Epoch 9/10 60000/60000 [==============================] - 129s 2ms/step - loss: 0.0611 - acc: 0.9811 - val_loss: 0.0987 - val_acc: 0.9727 Epoch 10/10 60000/60000 [==============================] - 136s 2ms/step - loss: 0.0533 - acc: 0.9837 - val_loss: 0.1070 - val_acc: 0.9718
<keras.callbacks.History at 0x7fdd95c86610>
# Więcej warstw, inna funkcja aktywacji
score = model3.evaluate(x_test, y_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.107020105763 Test accuracy: 0.9718
Przykład: 4-pikselowy aparat fotograficzny
YouTubeVideo('ILsA4nyG7I0', width=800, height=600)def generate_example(description):
variant = random.choice([1, -1])
if description == 's': # solid
return (np.array([[ 1.0, 1.0], [ 1.0, 1.0]]) if variant == 1 else
np.array([[-1.0, -1.0], [-1.0, -1.0]]))
elif description == 'v': # vertical
return (np.array([[ 1.0, -1.0], [ 1.0, -1.0]]) if variant == 1 else
np.array([[-1.0, 1.0], [-1.0, 1.0]]))
elif description == 'd': # diagonal
return (np.array([[ 1.0, -1.0], [-1.0, 1.0]]) if variant == 1 else
np.array([[-1.0, 1.0], [ 1.0, -1.0]]))
elif description == 'h': # horizontal
return (np.array([[ 1.0, 1.0], [-1.0, -1.0]]) if variant == 1 else
np.array([[-1.0, -1.0], [ 1.0, 1.0]]))
else:
return np.array([[random.uniform(-1, 1), random.uniform(-1, 1)],
[random.uniform(-1, 1), random.uniform(-1, 1)]])num_classes = 4
trainset_size = 4000
testset_size = 1000
y4_train = np.array([random.choice(['s', 'v', 'd', 'h']) for i in range(trainset_size)])
x4_train = np.array([generate_example(desc) for desc in y4_train])
y4_test = np.array([random.choice(['s', 'v', 'd', 'h']) for i in range(testset_size)])
x4_test = np.array([generate_example(desc) for desc in y4_test])draw_examples(x4_train[:7], captions=y4_train)s s d s h s v
x4_train = x4_train.reshape(trainset_size, 4)
x4_test = x4_test.reshape(testset_size, 4)
x4_train = x4_train.astype('float32')
x4_test = x4_test.astype('float32')
y4_train = np.array([{'s': 0, 'v': 1, 'd': 2, 'h': 3}[desc] for desc in y4_train])
y4_test = np.array([{'s': 0, 'v': 1, 'd': 2, 'h': 3}[desc] for desc in y4_test])
y4_train = keras.utils.to_categorical(y4_train, num_classes)
y4_test = keras.utils.to_categorical(y4_test, num_classes)model4 = Sequential()
model4.add(Dense(4, activation='tanh', input_shape=(4,)))
model4.add(Dense(4, activation='tanh'))
model4.add(Dense(8, activation='relu'))
model4.add(Dense(num_classes, activation='softmax'))
model4.summary()_________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_16 (Dense) (None, 4) 20 _________________________________________________________________ dense_17 (Dense) (None, 4) 20 _________________________________________________________________ dense_18 (Dense) (None, 8) 40 _________________________________________________________________ dense_19 (Dense) (None, 4) 36 ================================================================= Total params: 116 Trainable params: 116 Non-trainable params: 0 _________________________________________________________________
model4.layers[0].set_weights(
[np.array([[ 1.0, 0.0, 1.0, 0.0],
[ 0.0, 1.0, 0.0, 1.0],
[ 1.0, 0.0, -1.0, 0.0],
[ 0.0, 1.0, 0.0, -1.0]],
dtype=np.float32), np.array([0., 0., 0., 0.], dtype=np.float32)])
model4.layers[1].set_weights(
[np.array([[ 1.0, -1.0, 0.0, 0.0],
[ 1.0, 1.0, 0.0, 0.0],
[ 0.0, 0.0, 1.0, -1.0],
[ 0.0, 0.0, -1.0, -1.0]],
dtype=np.float32), np.array([0., 0., 0., 0.], dtype=np.float32)])
model4.layers[2].set_weights(
[np.array([[ 1.0, -1.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0],
[ 0.0, 0.0, 1.0, -1.0, 0.0, 0.0, 0.0, 0.0],
[ 0.0, 0.0, 0.0, 0.0, 1.0, -1.0, 0.0, 0.0],
[ 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 1.0, -1.0]],
dtype=np.float32), np.array([0., 0., 0., 0., 0., 0., 0., 0.], dtype=np.float32)])model4.layers[3].set_weights(
[np.array([[ 1.0, 0.0, 0.0, 0.0],
[ 1.0, 0.0, 0.0, 0.0],
[ 0.0, 1.0, 0.0, 0.0],
[ 0.0, 1.0, 0.0, 0.0],
[ 0.0, 0.0, 1.0, 0.0],
[ 0.0, 0.0, 1.0, 0.0],
[ 0.0, 0.0, 0.0, 1.0],
[ 0.0, 0.0, 0.0, 1.0]],
dtype=np.float32), np.array([0., 0., 0., 0.], dtype=np.float32)])
model4.compile(loss='categorical_crossentropy',
optimizer=Adagrad(),
metrics=['accuracy'])for layer in model4.layers:
print(layer.get_weights())[array([[ 1., 0., 1., 0.],
[ 0., 1., 0., 1.],
[ 1., 0., -1., 0.],
[ 0., 1., 0., -1.]], dtype=float32), array([ 0., 0., 0., 0.], dtype=float32)]
[array([[ 1., -1., 0., 0.],
[ 1., 1., 0., 0.],
[ 0., 0., 1., -1.],
[ 0., 0., -1., -1.]], dtype=float32), array([ 0., 0., 0., 0.], dtype=float32)]
[array([[ 1., -1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., -1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., -1., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., -1.]], dtype=float32), array([ 0., 0., 0., 0., 0., 0., 0., 0.], dtype=float32)]
[array([[ 1., 0., 0., 0.],
[ 1., 0., 0., 0.],
[ 0., 1., 0., 0.],
[ 0., 1., 0., 0.],
[ 0., 0., 1., 0.],
[ 0., 0., 1., 0.],
[ 0., 0., 0., 1.],
[ 0., 0., 0., 1.]], dtype=float32), array([ 0., 0., 0., 0.], dtype=float32)]
model4.predict([np.array([[1.0, 1.0], [-1.0, -1.0]]).reshape(1, 4)])array([[ 0.17831734, 0.17831734, 0.17831734, 0.46504799]], dtype=float32)
score = model4.evaluate(x4_test, y4_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.765614629269 Test accuracy: 1.0
model5 = Sequential()
model5.add(Dense(4, activation='tanh', input_shape=(4,)))
model5.add(Dense(4, activation='tanh'))
model5.add(Dense(8, activation='relu'))
model5.add(Dense(num_classes, activation='softmax'))
model5.compile(loss='categorical_crossentropy',
optimizer=RMSprop(),
metrics=['accuracy'])
model5.summary()_________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense_20 (Dense) (None, 4) 20 _________________________________________________________________ dense_21 (Dense) (None, 4) 20 _________________________________________________________________ dense_22 (Dense) (None, 8) 40 _________________________________________________________________ dense_23 (Dense) (None, 4) 36 ================================================================= Total params: 116 Trainable params: 116 Non-trainable params: 0 _________________________________________________________________
model5.fit(x4_train, y4_train, epochs=8, validation_data=(x4_test, y4_test))Train on 4000 samples, validate on 1000 samples Epoch 1/8 4000/4000 [==============================] - 0s - loss: 1.1352 - acc: 0.5507 - val_loss: 1.0160 - val_acc: 0.7330 Epoch 2/8 4000/4000 [==============================] - 0s - loss: 0.8918 - acc: 0.8722 - val_loss: 0.8094 - val_acc: 0.8580 Epoch 3/8 4000/4000 [==============================] - 0s - loss: 0.6966 - acc: 0.8810 - val_loss: 0.6283 - val_acc: 0.8580 Epoch 4/8 4000/4000 [==============================] - 0s - loss: 0.5284 - acc: 0.8810 - val_loss: 0.4697 - val_acc: 0.8580 Epoch 5/8 4000/4000 [==============================] - 0s - loss: 0.3797 - acc: 0.9022 - val_loss: 0.3312 - val_acc: 1.0000 Epoch 6/8 4000/4000 [==============================] - 0s - loss: 0.2555 - acc: 1.0000 - val_loss: 0.2166 - val_acc: 1.0000 Epoch 7/8 4000/4000 [==============================] - 0s - loss: 0.1612 - acc: 1.0000 - val_loss: 0.1318 - val_acc: 1.0000 Epoch 8/8 4000/4000 [==============================] - 0s - loss: 0.0939 - acc: 1.0000 - val_loss: 0.0732 - val_acc: 1.0000
<keras.callbacks.History at 0x7f4d34067510>
model5.predict([np.array([[1.0, 1.0], [-1.0, -1.0]]).reshape(1, 4)])array([[ 0.00708295, 0.00192736, 0.02899081, 0.96199888]], dtype=float32)
score = model5.evaluate(x4_test, y4_test, verbose=0)
print('Test loss: {}'.format(score[0]))
print('Test accuracy: {}'.format(score[1]))Test loss: 0.0731911802292 Test accuracy: 1.0
import contextlib
@contextlib.contextmanager
def printoptions(*args, **kwargs):
original = np.get_printoptions()
np.set_printoptions(*args, **kwargs)
try:
yield
finally:
np.set_printoptions(**original)with printoptions(precision=1, suppress=True):
for layer in model5.layers:
print(layer.get_weights())[array([[-0.2, -0.5, 0.8, 1. ],
[-0.9, 0.1, -0.8, 0.2],
[-0.2, 0.4, 0.1, -0.4],
[-0.8, 0.8, 1. , 0.3]], dtype=float32), array([ 0. , -0. , 0.1, -0.1], dtype=float32)]
[array([[-0.4, 0.9, -1.3, 1.7],
[-0.4, -0.7, 0.3, -0.3],
[ 0.8, -0.9, -1.1, -0.2],
[ 1.3, 0.5, 0.4, -0.2]], dtype=float32), array([-0. , -0. , 0.2, 0. ], dtype=float32)]
[array([[-1.6, 0.3, 0.3, -0.3, -1.1, 1.2, 0.7, -1. ],
[ 0.4, 1.3, -0.9, 0.8, -0.4, -0.7, -1.2, -1. ],
[ 0.6, 1. , 0.9, -1. , -1.1, -0.2, -0.4, -0.3],
[ 1.1, 0.1, -0.9, 1.3, -0.3, -0.2, 0.2, -0.4]], dtype=float32), array([-0. , 0.2, -0.1, 0. , -0.1, -0. , -0.1, 0.1], dtype=float32)]
[array([[ 0.6, -1.5, 1.3, -1.4],
[-0.4, -1.6, -0.3, 1.2],
[ 1.2, 1.1, -0.3, -1.5],
[ 0.6, 1.4, -1.5, -1.2],
[ 0.2, -1.3, -0.9, 0.8],
[ 0.6, -1.5, 0.8, -1. ],
[ 0.4, -1.3, 0.4, 0.3],
[-1.3, 0.5, -0.9, 0.8]], dtype=float32), array([-0.8, 0.7, 0.4, 0.1], dtype=float32)]
12.3. Odmiany metody gradientu prostego
- Batch gradient descent
- Stochastic gradient descent
- Mini-batch gradient descent
_Batch gradient descent
- Klasyczna wersja metody gradientu prostego
- Obliczamy gradient funkcji kosztu względem całego zbioru treningowego: $$ \theta := \theta - \alpha \cdot \nabla_\theta J(\theta) $$
- Dlatego może działać bardzo powoli
- Nie można dodawać nowych przykładów na bieżąco w trakcie trenowania modelu (_online learning)
_Stochastic gradient descent (SGD)
- Aktualizacja parametrów dla każdego przykładu: $$ \theta := \theta - \alpha \cdot \nabla_\theta , J ! \left( \theta, x^{(i)}, y^{(i)} \right) $$
- Dużo szybszy niż _batch gradient descent
- Można dodawać nowe przykłady na bieżąco w trakcie trenowania (_online learning)
_Stochastic gradient descent (SGD)
- Częsta aktualizacja parametrów z dużą wariancją:
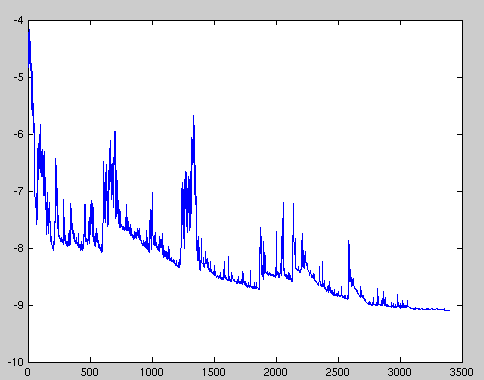
- Z jednej strony dzięki temu nie utyka w złych minimach lokalnych, ale z drugiej strony może „wyskoczyć” z dobrego minimum
_Mini-batch gradient descent
- Kompromis między _batch gradient descent i SGD $$ \theta := \theta - \alpha \cdot \nabla_\theta , J \left( \theta, x^{(i : i+n)}, y^{(i : i_n)} \right) $$
- Stabilniejsza zbieżność dzięki redukcji wariancji aktualizacji parametrów
- Szybszy niż klasyczny _batch gradient descent
- Typowa wielkość batcha: między 50 a 256 przykładów
Wady klasycznej metody gradientu prostego, czyli dlaczego potrzebujemy optymalizacji
- Trudno dobrać właściwą szybkość uczenia (_learning rate)
- Jedna ustalona wartość stałej uczenia się dla wszystkich parametrów
- Funkcja kosztu dla sieci neuronowych nie jest wypukła, więc uczenie może utknąć w złym minimum lokalnym lub punkcie siodłowym
12.4. Algorytmy optymalizacji metody gradientu
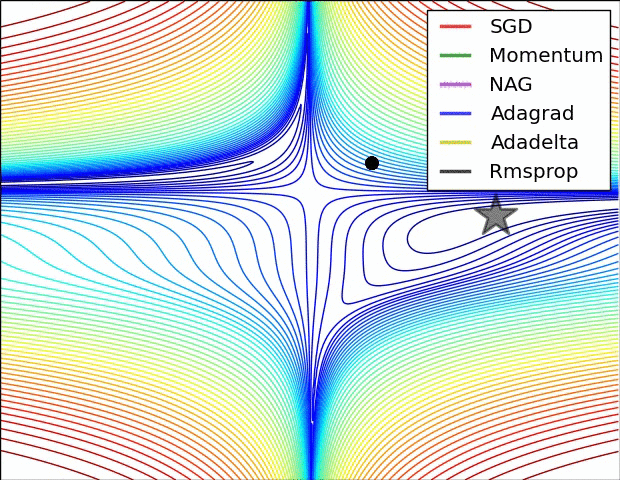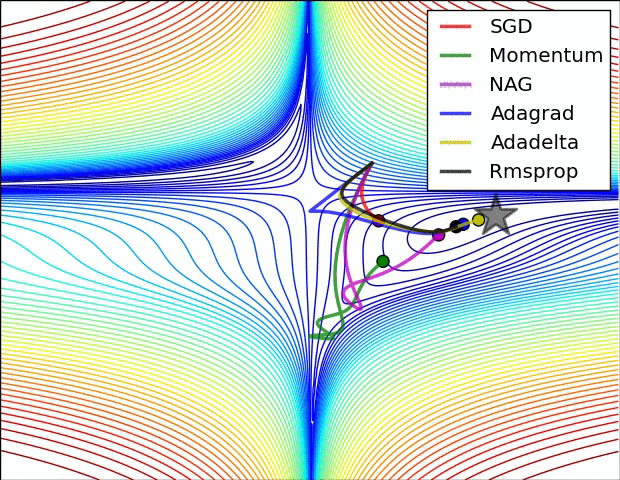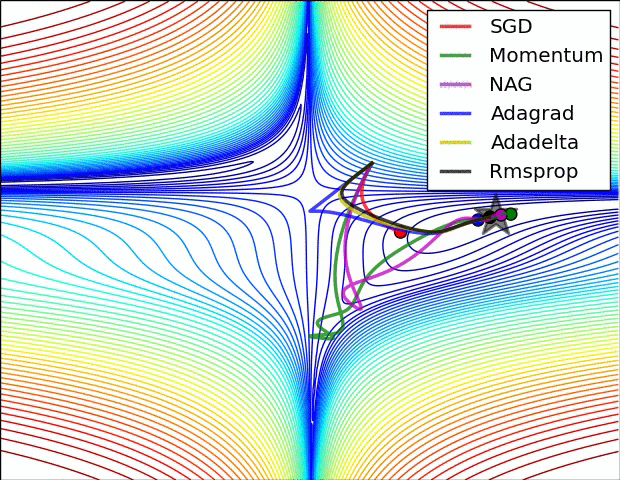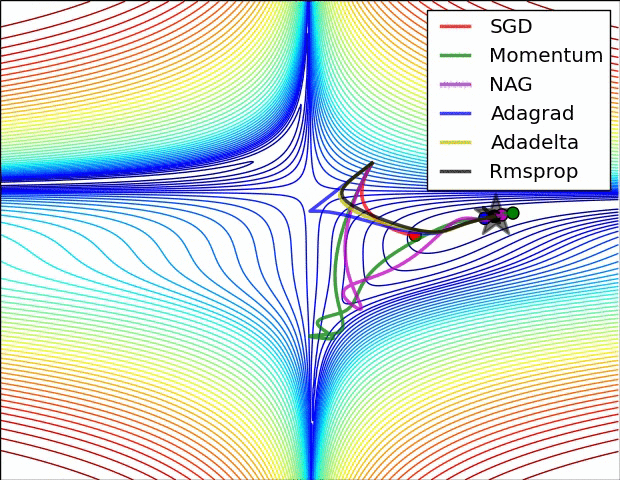
- Momentum
- Nesterov Accelerated Gradient
- Adagrad
- Adadelta
- RMSprop
- Adam
- Nadam
- AMSGrad
Momentum
- SGD źle radzi sobie w „wąwozach” funkcji kosztu
- Momentum rozwiązuje ten problem przez dodanie współczynnika $\gamma$, który można trakować jako „pęd” spadającej piłki: $$ v_t := \gamma , v_{t-1} + \alpha , \nabla_\theta J(\theta) $$ $$ \theta := \theta - v_t $$
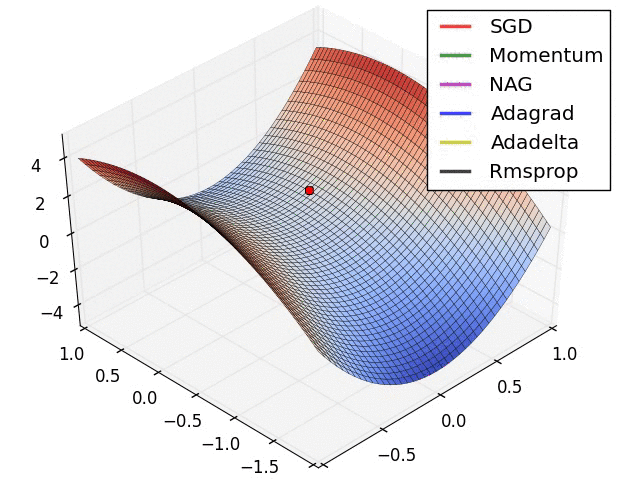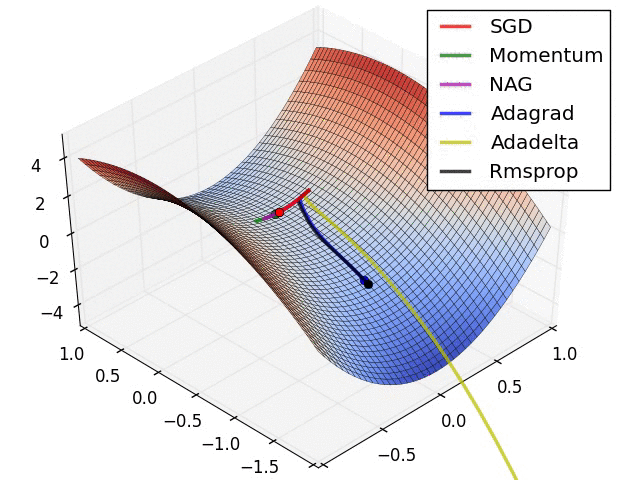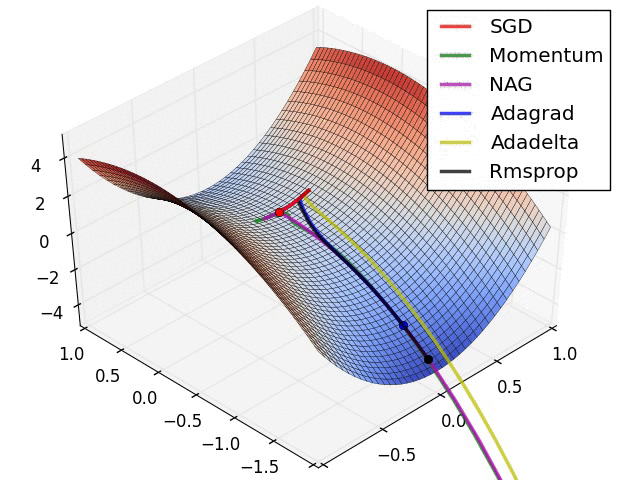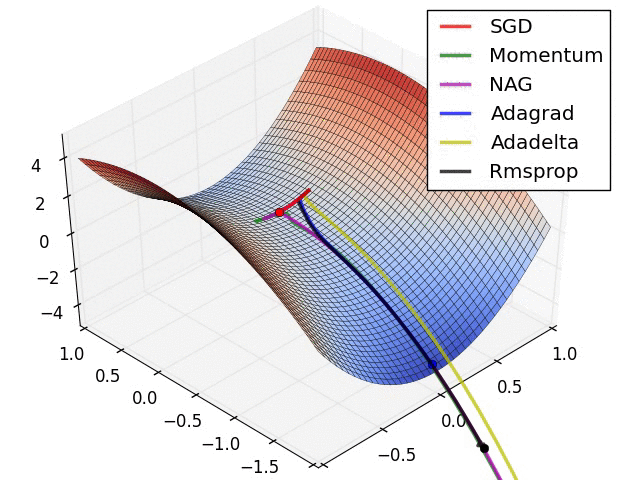
Przyspiesony gradient Nesterova (_Nesterov Accelerated Gradient, NAG)
- Momentum czasami powoduje niekontrolowane rozpędzanie się piłki, przez co staje się „mniej sterowna”
- Nesterov do piłki posiadającej pęd dodaje „hamulec”, który spowalnia piłkę przed wzniesieniem: $$ v_t := \gamma , v_{t-1} + \alpha , \nabla_\theta J(\theta - \gamma , v_{t-1}) $$ $$ \theta := \theta - v_t $$
Adagrad
- “Adaptive gradient”
- Adagrad dostosowuje współczynnik uczenia (_learning rate) do parametrów: zmniejsza go dla cech występujących częściej, a zwiększa dla występujących rzadziej
- Świetny do trenowania na rzadkich (_sparse) zbiorach danych
- Wada: współczynnik uczenia może czasami gwałtownie maleć
Adadelta i RMSprop
- Warianty algorytmu Adagrad, które radzą sobie z problemem gwałtownych zmian współczynnika uczenia
Adam
- “Adaptive moment estimation”
- Łączy zalety algorytmów RMSprop i Momentum
- Można go porównać do piłki mającej ciężar i opór
- Obecnie jeden z najpopularniejszych algorytmów optymalizacji
Nadam
- “Nesterov-accelerated adaptive moment estimation”
- Łączy zalety algorytmów Adam i Nesterov Accelerated Gradient
AMSGrad
- Wariant algorytmu Adam lepiej dostosowany do zadań takich jak rozpoznawanie obiektów czy tłumaczenie maszynowe