14 KiB
Raport z części indywidualnej - podprojektu
Tomasz Lech
Omówienie projektu
Celem projektu jest znalezienie najbardziej optymalnej drogi między zajętymi regałami a miejscami odbioru paczki. Projekt wykorzystuje wcześniej opracowany algorytm AStar, który jest opisany w pliku route-planning. Moduł podprojektu uruchamia się po uruchomieniu programu oraz naciśnięciu g na klawiaturze. Omawiany moduł genetyczny podprojektu w dalszej części raportu będzie się pojawiał w skrócie jako mdg.
Opis składowych elementów wykorzystanych w mdg
- Gen - jest to najmniejszy wykorzystywany obiekt, reprezentujący zajęty regał, kóry ma określony koszt do danego miejsca odbioru paczki.
- Chromosom - jest to uporządkowany zbiór Genów, który reprezentuje kolejność odbioru paczek, końcowa długość wynika z ilości paczek na magazynie.
- Populacja - jest to zbiór chromosomów.
- Funkcja fitness - funkcja obliczająca całkowity koszt chromosomu.
- Selekcja - składowa odpowiedzialna za wybór najlepszych chromosomów z pośród populacji.
- Crossover - składowa odpowiedzialna za generowanie nowej populacji uwzględniając współczynnik mutacji, wielkość dziedziczonego fragmentu oraz otrzymane podczas selekcji chromosomy.
Dane wejściowe
Po uruchomieniu programu:
- generowanie losowo rozmieszczonych paczek na regałach - po naciśnięciu r na klawiaturze.
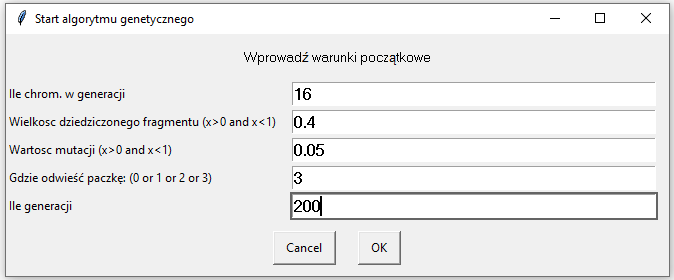
Po naciśnięciu g na klawiaturze pojawia się okno zbierające dane wejśćiowe:
- Warotść definiująca liość chromosomów w każdej populacji
- Wardośc definiująca wielkość fragmentu genu, kótry będzie dziedziczony przy tworzeniu nowych chromosomów
- Wartośc definiująca jaki fragment zmutować
- Wartość definiująca do jakiego miejsca odwieść paczkę. (1 - miejsce oddania paczki po lewej stronie, 2 - miejsce oddania paczki po prawej stronie,
3 - miejsce oddania paczki wybierane na podstawie kosztu) - Wartość definiująca ile stworzyć generacji w trakcie działania modułu
Integracja
W pliku program.py
Uruchomienie mdg:
if event.key == pygame.K_g:
start(self.data,self.wheel)
Po zakończeniu algorytmu, uruchamiamy moduł, który rozwiezie paczki do miejsca odbioru:
for gen in self.data.best[0]:
if(gen.unboxWczesniejszegoGenu == None):
kordStartowy = (self.wheel.ns, self.wheel.we)
else:
kordStartowy = self.data.unbox[gen.unboxWczesniejszegoGenu]
zbierzBox(gen,self.data, self.moves, kordStartowy)
W pliku genetyczne.py
def start(data, wheel):
ileGeneracji = 20
ileWPopulacji = 16
fragment = 0.5
mutacja = 0.05
unbox = 3
data.kordyWozka = (wheel.ns, wheel.we)
data.jakLiczycKoszt = unbox
randomPopulation = genRandomPopulation(data, ileWPopulacji)
for i in range(ileGeneracji):
if i == 0:
best2 = dwieNajlepsze(randomPopulation, data)
else:
x = genPopulacje(data,best2[0], best2[1], ileWPopulacji, fragment, mutacja)
best2 = dwieNajlepsze(x, data)
del x
data.histZmian.append(data.best[1])
rysujWykres(data, ileGeneracji, 0, 2000)
W celu modyfikacji danych wejściowych należy zmienić wartości zmiennych, pamiętając o podanych powyżej ograniczeniach.
Powyżej fragment kodu reprezentujący działanie pętli, której iteracje odpowiadają tworzeniom nowych generacji.
Sposób działania algorytmu:
graph TD
A[<center> Generowanie <br/> losowego <br/>chromosomu<center/>]
B[<center> Generowanie <br/> losowej <br/> populacji <center/>]
C[<center> Selekcyjny <br/> wybór najlepszych chromosomów <br/>z pośród populacji <center/>]
D[Generowanie nowej populacji z podanych chromosomów]
E[Ilość generacji]
A --> B
B --> C
C --> D
D --> E
E --> C
Implementacja
Generowanie losowego chromosomu
W pliku Gene.py
Klasa Gene:
class Gene:
def __init__(self):
self.kordy = None
self.unbox1 = None
self.unbox2 = None
self.unboxWczesniejszegoGenu = None
self.kordyUnboxa = None
Odpowiednio:
- kordy - krotka z koordynatami regału
- unbox1 - koszt potrzebny do przejazdu z miejsca regału do miejsca oddania paczki po lewej stronie mapy
- unbox2 - koszty potrzebny do przejazdu z miejsca regału do miejsca oddania paczki po prawej stronie mapy
- unboxWczesniejszegoGenu - wartość (0 lub 1) która definiuje z jakiego miejsca oddania paczki jechał wózek do regału reprezentowanego przez ten gen
- kordyUnboxa - koordynaty miejsca oddania paczki do którego będzie jechać wózek
Od tego momentu miejsce oddania paczki będzie określane jako unbox
W pliku genetyczne.py
def generateGeny(data):
geny = []
zajeteRegaly = data.zajeteRegaly[:]
for r in zajeteRegaly:
g = Gene()
g.kordy = r
g.unbox1 = policzCost(data.astarMap,r,data.unbox[0])
if(len(data.unbox) > 1):
g.unbox2 = policzCost(data.astarMap,r,data.unbox[1])
geny.append(g)
return geny
def genRandomChromosome(data):
chromosome = generateGeny(data)
random.shuffle(chromosome)
unboxLastGen = None
for gen in chromosome:
gen.unboxWczesniejszegoGenu = unboxLastGen
krotkaKosztJakiUnbox = wybierzUnbox(gen, data.jakLiczycKoszt)
unboxLastGen = krotkaKosztJakiUnbox[1]
gen.kordyUnboxa = data.unbox[krotkaKosztJakiUnbox[1]]
return chromosome
Odpowiednio:
- Funkcja generateGeny generuje oraz oblicza wartości unboxów dla danego regału oraz zwraca je jako listę genów
- Funkcja genRandomChromosome losowo miesza wygenerowane geny oraz dla podanej wartości unbox (podanej przy uruchomieniu programu) zapisuje w genach wartości odpowiadające koodrynatom unboxa oraz z jakiego unboxa wózek przyjedzie. W przypadku pierwszego genu, do którego wózek będzie jechać z określonego miejsca ta wartość pozostaje None. Funkcja zwraca spójny chromosom.
Generowanie Losowej populacji
W pliku genetyczne.py
def genRandomPopulation(data, ileWPopulacji):
populacja = []
for i in range(ileWPopulacji):
populacja.append(genRandomChromosome(data))
return populacja
Odpowiednio:
- Dla podanej wartości ileWPopulacji funkcja generuje losową populację, wykonując tyle iteracji ile wynosi wartość.
Selekcyjny wybór najlepszych chromosomów z pośród populacji na podstawie funkcji fitness
W pliku genetyczne.py
def fitness(chromosome, data):
koszt = 0
unboxPoprzedniegoGenu = None
for item, gen in enumerate(chromosome):
if(item == 0):
koszt += policzCost(data.astarMap, data.kordyWozka, gen.kordy)
krotkaKosztJakiUnbox = wybierzUnbox(gen, data.jakLiczycKoszt)
koszt += krotkaKosztJakiUnbox[0]
unboxPoprzedniegoGenu = krotkaKosztJakiUnbox[1]
else:
if unboxPoprzedniegoGenu == 0:
koszt += gen.unbox1
elif unboxPoprzedniegoGenu == 1:
koszt += gen.unbox2
krotkaKosztJakiUnbox = wybierzUnbox(gen, data.jakLiczycKoszt)
koszt += krotkaKosztJakiUnbox[0]
unboxPoprzedniegoGenu = krotkaKosztJakiUnbox[1]
return koszt
Odpowiednio:
-
Zmienna koszt jest sumą całkowitą kosztów przejechania trasy.
-
Pętla for iteruje się tyle razy ile jest genów w chromosomie.
-
W pierwszej iteracji koszt jest liczony dla pierwszego genu w chromosomie wywołując AStar, z pozycji początkowej wózka, do miejsca regału.
-
Dla reszty iteracji jest sprawdzane do którego unboxa będzie jechać wózek, i taka wartość kosztu jest dodawana co całkowitej sumy oraz koszt przejechania od unboxa poprzedniego genu do regału (zmienna unboxPoprzedniegoGenu)
def dwieNajlepsze(populacja, data):
tmpPopulacja = populacja[:] chromFitness = []for chrom in populacja: chromFitness.append(fitness(chrom,data)) bestValue = min(chromFitness) bestChromIndex = chromFitness.index(bestValue) pierwsza = tmpPopulacja[bestChromIndex] if (data.best == None): data.best = (pierwsza[:],bestValue) elif(data.best[1] > bestValue): data.best = (pierwsza[:],bestValue) data.doWykresu.append(bestValue) tmpPopulacja.pop(bestChromIndex) chromFitness.pop(bestChromIndex) bestValue = min(chromFitness) bestChromIndex = chromFitness.index(bestValue) druga = tmpPopulacja[bestChromIndex] tmpPopulacja.pop(bestChromIndex) chromFitness.pop(bestChromIndex) return (pierwsza, druga)
Funkcja selekcji dla której odpowiednio:
- W pierwszej pętli for tworzy się lista chromFitness przetrzymująca wartości kosztów dla danego chromosomu. Wartości w chromFitness odpowiadają chromosomom na tych samych indeksach w liście populacja.
- Zmienna bestValue reprezentuje najlepszy koszt z danej populacji
- Zmienna pierwsza reprezentuje chromosom o najkorzystniejszym koszcie.
- Zmienna druga reprezentuje chromosom o drugim co do wartości najkorzystniejszym koszcie.
- W zmiennej best klasy obiektu data zapisywana jest krotka odpowiednio (chromosom,koszt) najlepszego chromosomu.
- Funkcja zwraca krotkę z dwoma najlepszymi chromosomami w populacji.
Generowanie nowej populacji - Crossover
W pliku genetyczne.py
def crossover(data,pierwszy, drugi, fragmentLiczba, wspMutacji):
ileWChrom = len(pierwszy)
tmp = random.randint(0, ileWChrom-fragmentLiczba)
kordyFragment = (tmp,tmp+fragmentLiczba)
nowyChrom = [Gene() for q in range(ileWChrom)]
iterator = kordyFragment[1]
pomIterator = kordyFragment[1]
usedKordy = []
for i in range(kordyFragment[0],kordyFragment[1]):
nowyChrom[i].kordy = pierwszy[i].kordy
nowyChrom[i].unbox1 = pierwszy[i].unbox1
nowyChrom[i].unbox2 = pierwszy[i].unbox2
usedKordy.append(pierwszy[i].kordy)
for x in range(ileWChrom):
if(iterator > ileWChrom - 1):
iterator = 0
if(pomIterator > ileWChrom - 1):
pomIterator = 0
if(nowyChrom[iterator].kordy == None and drugi[pomIterator].kordy not in usedKordy):
nowyChrom[iterator].kordy = drugi[pomIterator].kordy
nowyChrom[iterator].kordy = drugi[pomIterator].kordy
nowyChrom[iterator].unbox1 = drugi[pomIterator].unbox1
nowyChrom[iterator].unbox2 = drugi[pomIterator].unbox2
iterator += 1
pomIterator += 1
else:
pomIterator +=1
nowyChrom = mutate(wspMutacji, nowyChrom)
unboxLastGen = None
for gen in nowyChrom:
gen.unboxWczesniejszegoGenu = unboxLastGen
krotkaKosztJakiUnbox = wybierzUnbox(gen, data.jakLiczycKoszt)
unboxLastGen = krotkaKosztJakiUnbox[1]
gen.kordyUnboxa = data.unbox[krotkaKosztJakiUnbox[1]]
return nowyChrom
Odpowiednio:
- Dane wejściowe są to:
- pierwszy, drugi - wybrane najkorzystniejsze chromosomy, z których ma powstać nowy chromosom
- fragmentLiczba - jest to liczba reprezentująca jaki fragment z pierwszego chromosomu zostanie bezpośrednio skopiowany do nowego chromosomu, ten fragment jest wybierany losowo spośród chromosomu natomiast jego długość jest określona procentowo i zależy od podanej wartości (oraz ilości genów w chromosomoie)
- wspMutacji - jest to liczba reprezentująca jak wiele par w chromosomie zostanie zamienionych miejscami.
- Zmienne pomocnicze:
- iterator, pomIterator - w pierwszych dwóch instrukcjach warunkowych jest pilnowane aby iterując się nie przekroczyły dopuszczalnej wartości (odpowiadają one indeksom w kolejce). Iterator jest indeksem w nowym, tworzonym chromosomie. pomIterator jest indeksem który przechodzi przez drugi podany chromosom.
- lista usedKordy - do niej są dodawane koordynaty genów, które zostały skopiowane z pierwszego chromosomu, aby geny o tych samych koordynatach z drugiego chromosomu nie zostały zapisane w nowym chromosomie.
- Następuje skopiowanie fagmentu z pierwszego chromosomu, w pierwszej pętli for wykonuje się przepisanie wartości do powstającego chromosomu. W drugiej pętli for następuje przepisanie pozostałych wartości z drugiego chromosomu do powstającego chromosomu.
- Po przepisaniu wartości według wspMutacji jest dokonywana zamiana genów w nowym chromosomie.
- Ostatnia pętla for łączy geny ze sobą (zapisując unbox poprzedniego genu)
W pliku genetyczne.py
def genPopulacje(data,pierwszy, drugi, ileWPopulacji, fragmentLiczba, wspMutacji):
ileWChrom = len(pierwszy)
fragment = round(fragmentLiczba*ileWChrom)
if(fragment == 1):
fragment +=1
nowaPopulacja = []
for i in range(ileWPopulacji):
nowaPopulacja.append(crossover(data,pierwszy,drugi,fragment, wspMutacji))
return nowaPopulacja
Odpowiednio:
- W pętli for tworzone są nowe chromosomy z pierwszego oraz drugiego najlepszego chromosomu z poprzedniej generacji.
- Nowe chromosomy zapisywane są do nowaPopulacja
- Z powstałej populacji na nowo selekcjonowane są dwa najlepsze, z których będą powstawać nowe populacje w zależności od wartości podanych generacji.
Dalsze działanie programu
Po zakończeniu mdg wózek zaczyna rozwozić paczki do miejsc oddania paczki.