forked from s444399/AI
Compare commits
26 Commits
| Author | SHA1 | Date | |
|---|---|---|---|
| c16a984b40 | |||
| 86d2bb2d0a | |||
| 05ef2f7ae2 | |||
| 27bf24d368 | |||
| 688d1483fb | |||
| bbda516f85 | |||
| 1d533cd819 | |||
| 9eeb0c58b6 | |||
| e9cf805873 | |||
| 97819433f6 | |||
| 53824177ca | |||
| 92b27ca85c | |||
| 457de041ac | |||
| af22640d83 | |||
| 45c67cb8e6 | |||
| e2be2d2721 | |||
| bba60512ae | |||
| b0988bf568 | |||
| cef5c23ed0 | |||
| 2d98954114 | |||
| c2babacae5 | |||
| c960a23fa0 | |||
| 1ee3f7dae3 | |||
| b4885a77f3 | |||
| f9d5c017d7 | |||
| bb9c559b4e |
53
LechT.md
53
LechT.md
@ -4,12 +4,12 @@
|
||||
### Omówienie projektu
|
||||
|
||||
|
||||
Celem projektu jest znalezienie najoptymalniejszej drogi między zajętymi regałami a miejscami odbioru paczki.
|
||||
Celem projektu jest znalezienie najbardziej optymalnej drogi między zajętymi regałami a miejscami odbioru paczki.
|
||||
Projekt wykorzystuje wcześniej opracowany algorytm AStar, który jest opisany w pliku [route-planning](https://git.wmi.amu.edu.pl/s444399/AI/src/master/route-planning.md).
|
||||
Moduł podprojektu uruchamia się po uruchomieniu programu oraz kliknięciu **g** na klawiaturze. Omawiany moduł genetyczny podprojektu w dalszej części raportu będzie się pojawiał w skrócie jako **mdg**.
|
||||
Moduł podprojektu uruchamia się po uruchomieniu programu oraz naciśnięciu **g** na klawiaturze. Omawiany moduł genetyczny podprojektu w dalszej części raportu będzie się pojawiał w skrócie jako **mdg**.
|
||||
|
||||
### Opis składowych elementów wykorzystanych w **mdg**
|
||||
* Gen - jest to najmniejszy wykorzystywany obiekt, reprezentujący zajęty regał, kóry ma określony koszt do danego miejsca odbioru
|
||||
* Gen - jest to najmniejszy wykorzystywany obiekt, reprezentujący zajęty regał, kóry ma określony koszt do danego miejsca odbioru paczki.
|
||||
* Chromosom - jest to uporządkowany zbiór Genów, który reprezentuje kolejność odbioru paczek, końcowa długość wynika z ilości paczek na magazynie.
|
||||
* Populacja - jest to zbiór chromosomów.
|
||||
* Funkcja fitness - funkcja obliczająca całkowity koszt chromosomu.
|
||||
@ -20,22 +20,24 @@ Moduł podprojektu uruchamia się po uruchomieniu programu oraz kliknięciu **g*
|
||||
|
||||
### Dane wejściowe
|
||||
|
||||
Podane przez urzydkownika przed uruchomieniem programu:
|
||||
|
||||
* ileGeneracji - wartość, która definiuje ile generacji ma się wykonać po uruchomieniu modułu **mdg**,
|
||||
* ileWPopulacji - wartość, która definiuje ile chromosomów ma się znajdować w Populacji
|
||||
* fragment - wartość z zakresu (0,1), która względnie do długości chromosomu określa fragment, który będzie dziedziczony, przy tworzeniu nowego chromosomu.
|
||||
* mutacja - wartość z zakresu (0,1), która określa jaka część nowo tworzonego chromosomu, po dziedziczeniu, ma zostać losowo zmieniona.
|
||||
* unbox - wartość określająca do jakiego miejsca odbioru ma się kierować wózek
|
||||
<br/>
|
||||
0 - losowe miejsce odbioru <br/>
|
||||
1 - miejsce odbioru tylko po lewej stronie mapy <br/>
|
||||
2 - miejsce odbioru tylko po prawej stronie mapy <br/>
|
||||
3 - miejsce odbioru wybierane korzystniej na podstawie kosztu
|
||||
<br/><br/>
|
||||
Po uruchomieniu programu:
|
||||
|
||||
* generowanie losowo rozmieszczonych paczek na regałach - za przycisku **r** na klawiaturze.
|
||||
* generowanie losowo rozmieszczonych paczek na regałach - po naciśnięciu **r** na klawiaturze.
|
||||
|
||||
Po naciśnięciu **g** na klawiaturze pojawia się okno zbierające dane wejśćiowe:
|
||||
|
||||
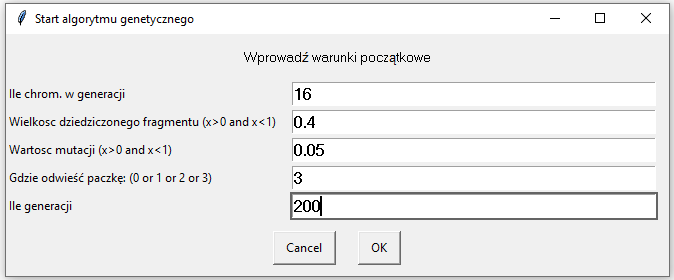
|
||||
|
||||
<ol>
|
||||
<li>Warotść definiująca liość chromosomów w każdej populacji</li>
|
||||
<li>Wardośc definiująca wielkość fragmentu genu, kótry będzie dziedziczony przy tworzeniu nowych chromosomów</li>
|
||||
<li>Wartośc definiująca jaki fragment zmutować</li>
|
||||
<li>Wartość definiująca do jakiego miejsca odwieść paczkę. (1 - miejsce oddania paczki po lewej stronie, 2 - miejsce oddania paczki po prawej stronie, <br/> 3 - miejsce oddania paczki wybierane na podstawie kosztu) </li>
|
||||
<li>Wartość definiująca ile stworzyć generacji w trakcie działania modułu</li>
|
||||
</ol>
|
||||
odbioru wybierane korzystniej na podstawie kosztu
|
||||
<br/><br/>
|
||||
|
||||
|
||||
|
||||
### Integracja
|
||||
@ -48,7 +50,7 @@ Uruchomienie **mdg**:
|
||||
if event.key == pygame.K_g:
|
||||
start(self.data,self.wheel)
|
||||
|
||||
Po zakończeniu algorytmu, uruchaminy modul który rozwiezie paczki do miejsca odbioru:
|
||||
Po zakończeniu algorytmu, uruchamiamy moduł, który rozwiezie paczki do miejsca odbioru:
|
||||
|
||||
for gen in self.data.best[0]:
|
||||
if(gen.unboxWczesniejszegoGenu == None):
|
||||
@ -85,7 +87,7 @@ Po zakończeniu algorytmu, uruchaminy modul który rozwiezie paczki do miejsca o
|
||||
|
||||
rysujWykres(data, ileGeneracji, 0, 2000)
|
||||
|
||||
W celu modyfikacji danych wejściowych należy zmienić wartości zmiennych, pamiętając o podanych powyrzej ograniczeniach.
|
||||
W celu modyfikacji danych wejściowych należy zmienić wartości zmiennych, pamiętając o podanych powyżej ograniczeniach.
|
||||
|
||||
Powyżej fragment kodu reprezentujący działanie pętli, której iteracje odpowiadają tworzeniom nowych generacji.
|
||||
|
||||
@ -107,6 +109,7 @@ E[Ilość generacji]
|
||||
E --> C
|
||||
```
|
||||
### Implementacja
|
||||
|
||||
#### Generowanie losowego chromosomu
|
||||
*W pliku Gene.py*
|
||||
|
||||
@ -168,7 +171,7 @@ Odpowiednio:
|
||||
populacja.append(genRandomChromosome(data))
|
||||
return populacja
|
||||
Odpowiednio:
|
||||
* Dla podanej wartości *ileWPopulacji* funkcja generuje losową populację wykorzystując metodę losowego chromosomu, wykonując tyle iteracji ile wynosi wartość.
|
||||
* Dla podanej wartości *ileWPopulacji* funkcja generuje losową populację, wykonując tyle iteracji ile wynosi wartość.
|
||||
|
||||
#### Selekcyjny wybór najlepszych chromosomów z pośród populacji na podstawie funkcji fitness
|
||||
*W pliku genetyczne.py*
|
||||
@ -200,10 +203,12 @@ Odpowiednio:
|
||||
Odpowiednio:
|
||||
* Zmienna *koszt* jest sumą całkowitą kosztów przejechania trasy.
|
||||
* Pętla *for* iteruje się tyle razy ile jest genów w chromosomie.
|
||||
* W pierwszej iteracji koszt jest liczony dla pierwszego genu w chromosomie wywołując AStar, z pozycji początkowej wózka do miejsca regału.
|
||||
* W pierwszej iteracji koszt jest liczony dla pierwszego genu w chromosomie wywołując AStar, z pozycji początkowej wózka, do miejsca regału.
|
||||
* Dla reszty iteracji jest sprawdzane do którego unboxa będzie jechać wózek, i taka wartość kosztu jest dodawana co całkowitej sumy oraz koszt przejechania od unboxa poprzedniego genu do regału (zmienna *unboxPoprzedniegoGenu*)
|
||||
|
||||
|
||||
|
||||
|
||||
def dwieNajlepsze(populacja, data):
|
||||
tmpPopulacja = populacja[:]
|
||||
chromFitness = []
|
||||
@ -289,7 +294,7 @@ Odpowiednio:
|
||||
* *fragmentLiczba* - jest to liczba reprezentująca jaki fragment z **pierwszego** chromosomu zostanie bezpośrednio skopiowany do nowego chromosomu, ten fragment jest wybierany losowo spośród chromosomu natomiast jego długość jest określona procentowo i zależy od podanej wartości (oraz ilości genów w chromosomoie)
|
||||
* *wspMutacji* - jest to liczba reprezentująca jak wiele par w chromosomie zostanie zamienionych miejscami.
|
||||
* Zmienne pomocnicze:
|
||||
* *iterator*, *pomIterator* - w pierwszych dwóch instrukcjach warunkowych jest pilnowane aby iterując się nie przekroczyły dopuszczalnej wartości (odpowiadają one indeksom w kolejce). *Iterato* jest indeksem w nowym, tworzonym chromosomie. *pomIterator* jest indeksem który przechodzi przez **drugi** podany chromosom.
|
||||
* *iterator*, *pomIterator* - w pierwszych dwóch instrukcjach warunkowych jest pilnowane aby iterując się nie przekroczyły dopuszczalnej wartości (odpowiadają one indeksom w kolejce). *Iterator* jest indeksem w nowym, tworzonym chromosomie. *pomIterator* jest indeksem który przechodzi przez **drugi** podany chromosom.
|
||||
* lista *usedKordy* - do niej są dodawane koordynaty genów, które zostały skopiowane z **pierwszego** chromosomu, aby geny o tych samych koordynatach z **drugiego** chromosomu nie zostały zapisane w nowym chromosomie.
|
||||
* Następuje skopiowanie fagmentu z **pierwszego** chromosomu, w pierwszej pętli *for* wykonuje się przepisanie wartości do powstającego chromosomu. W drugiej pętli *for* następuje przepisanie pozostałych wartości z **drugiego** chromosomu do powstającego chromosomu.
|
||||
* Po przepisaniu wartości według wspMutacji jest dokonywana zamiana genów w nowym chromosomie.
|
||||
@ -315,6 +320,4 @@ Odpowiednio:
|
||||
|
||||
### Dalsze działanie programu
|
||||
|
||||
Po wykonaniu iteracji uruchamia się okienko pokazujące wykres najlepszych wag otrzymywanych w danej populacji.
|
||||
|
||||
Po zamknięciu okienka wózek zaczyna rozwozić paczki do miejsc oddania paczki.
|
||||
Po zakończeniu **mdg** wózek zaczyna rozwozić paczki do miejsc oddania paczki.
|
||||
BIN
demo/7.png
Normal file
BIN
demo/7.png
Normal file
Binary file not shown.
|
After Width: | Height: | Size: 10 KiB |
41
final-evaluation.md
Normal file
41
final-evaluation.md
Normal file
@ -0,0 +1,41 @@
|
||||
## Raport finalny - Inteligentny Wózek Widłowy
|
||||
|
||||
### Uruchamianie
|
||||
|
||||
Po uruchomieniu programu musimy podać jakiej wielkości kratę chcemy uzyskać, a następnie jakie ilości których regałów chcemy posiadać na magazynie.
|
||||
|
||||

|
||||
|
||||
Gdy ukaże nam się okno programu możemy poruszać się wózkiem po magazynie. Używamy do tego strzałek. Naciśnięcie przycisku **r** na klawiaturze rozłoży paczki losowo na półkach.
|
||||
|
||||

|
||||
|
||||
## Podprojekty indywidualne
|
||||
|
||||
### Rozpoznawanie paczki
|
||||
|
||||
**Autor:** Michał Kijowski <br>
|
||||
**Wykorzystana metoda uczenia:** Sieci neuronowe.
|
||||
|
||||
Pełne omówienie podprojektu znajduje się w pliku [KijowskiM.md](https://git.wmi.amu.edu.pl/s444399/AI/src/master/KijowskiM.md).
|
||||
|
||||
Po kliknięciu w pole programu należy wybrać plik graficzny reprezentujący wygląd paczki. Algorytm za pomocą sieci neuronowych na podstawie odpowiednich piktogramów decyduje jakiego rodzaju jest paczka - krucha, niebezpieczna, radioaktywna, łatwopalna lub zwykła. Następnie informacja ta przekazywana jest do kolejnego podprojektu w celu odpowiedniego rozmieszczenia paczki.
|
||||
|
||||
### Rozmieszczenie w magazynie
|
||||
|
||||
**Autor:** Weronika Gorący <br>
|
||||
**Wykorzystana metoda uczenia:** Drzewa decyzyjne.
|
||||
|
||||
Pełne omówienie podprojektu znajduje się w pliku [WGoracy.md](https://git.wmi.amu.edu.pl/s444399/AI/src/master/WGoracy.md).
|
||||
|
||||
Podprojekt zostaje uruchomiony bezpośrednio po zakończeniu wykonywania poprzedniego podprojektu. Po otrzymaniu informacji o paczce, wózek za pomocą drzewa decyzyjnego wybiera najlepszą możliwą pustą półkę na której można umieścić paczkę. W przypadku gdy wszystkie półki danego rodzaju są zajęte, paczka zostaje umieszczona na innej dostępnej półce.
|
||||
|
||||
### Odbiór paczki
|
||||
|
||||
**Autor:** Tomasz Lech <br>
|
||||
**Wykorzystana metoda uczenia:** Algorytmy genetyczne.
|
||||
|
||||
Pełne omówienie podprojektu znajduje się w pliku [LechT.md](https://git.wmi.amu.edu.pl/s444399/AI/src/master/LechT.md).
|
||||
|
||||
Moduł podprojektu uruchamia się po naciśnięciu przycisku **g** na klawiaturze. Pojawi się okno w którym definiujemy odpowiednie wartości potrzebne do uruchomienia modułu.
|
||||
Podprojekt za pomocą algorytmu genetycznego wybiera najbardziej optymalną drogę pomiędzy zajętymi półkami a miejscami odbioru paczek. A następnie wózek zaczyna rozwozić paczki do miejsc oddania paczki.
|
||||
Loading…
Reference in New Issue
Block a user