3.5 MiB
Regresja jądrowa
Karolina Oparczyk, Tomasz Grzybowski, Jan Nowak
Regresja jądrowa używana jest jako funkcja wagi do opracowania modelu regresji nieparametrycznej. Nadaje ona niektórym elementom zbioru większą "wagę", która ma wpływ na ostateczny wynik.
Można ją porównać do rysowania krzywej na wykresie punktowym tak, aby była jak najlepiej do nich dopasowana.
Właściwości regresji jądrowej:
Do implementacji regresji jądrowej można użyć wielu różnych jąder. Przykłady użyte w projekcie:
- jądro Gaussa \begin{equation} K(x) = \frac1{h\sqrt{2\pi}}e^{-\frac12(\frac{x - x_i}h)^2} \end{equation}
import KernelRegression
import plotly.express as px
import numpy as np
kernel_x = np.arange(-4,4,0.1)
col = KernelRegression.kernel_function(1, kernel_x, 0)
px.line(x=kernel_x, y=col, title='Funkcja jądrowa Gaussa')- jądro Epanechnikova \begin{equation} K(x) = (\frac34)(1-(\frac{x - x_i}h)^2) \text{ dla } {|x|\leq1} \end{equation}
kernel_x = np.arange(-2,2,0.1)
col = KernelRegression.epanechnikov_list(1, kernel_x, 0)
px.line(x=kernel_x, y=col, title='Funkcja jądrowa Epanechnikova')Istotne znaczenie ma nie tylko dobór jądra, ale również parametru wygładzania, czyli szerokości okna. W zależności od niego, punkty są grupowane i dla każdej grupy wyliczana jest wartość funkcji. Jeśli okno będzie zbyt szerokie, funkcja będzie bardziej przypominała prostą (under-fitting). Natomiast jeśli będzie zbyt wąskie, funkcja będzie za bardzo "skakać" (over-fitting).
Wyliczenie wartości funkcji polega na wzięciu średniej ważonej z $y_{i}$
dla takich $x_{i}$, które znajdują się blisko x, dla którego wyznaczamy wartość. Wagi przy $y_{i}$ dla x sumują się do 1 i są wyższe, kiedy $x_{i}$ jest bliżej x oraz niższe w przeciwnym przypadku.
\begin{equation} Y=w_1*x_1+w_2*x_2+ \text{...} +w_n*x_n \end{equation}
import ipywidgets as widgets
import plotly.graph_objs as go
import pandas as pd
fires_thefts = pd.read_csv('fires_thefts.csv', names=['x','y'])
fires_thefts=fires_thefts.sort_values('x')
X = np.array(fires_thefts.x)
Y = np.array(fires_thefts.y)
dropdown_bw = widgets.Dropdown(options=[1, 2, 3, 4, 5, 6, 7, 8, 9, 10], value=1, description='Szerokość okna')
def interactive_kernel(bw_manual):
Y_pred_gauss = KernelRegression.ker_reg(X, Y, bw_manual, 'gauss')
Y_pred_epanechnikov = KernelRegression.ker_reg(X, Y, bw_manual, 'epanechnikov')
fig = px.scatter(x=X,y=Y)
fig.add_trace(go.Scatter(x=X, y=np.array(Y_pred_gauss), name='Gauss', mode='lines'))
fig.add_trace(go.Scatter(x=X, y=np.array(Y_pred_epanechnikov), name='Epanechnikov', mode='lines'))
fig.show()
# kernel regression
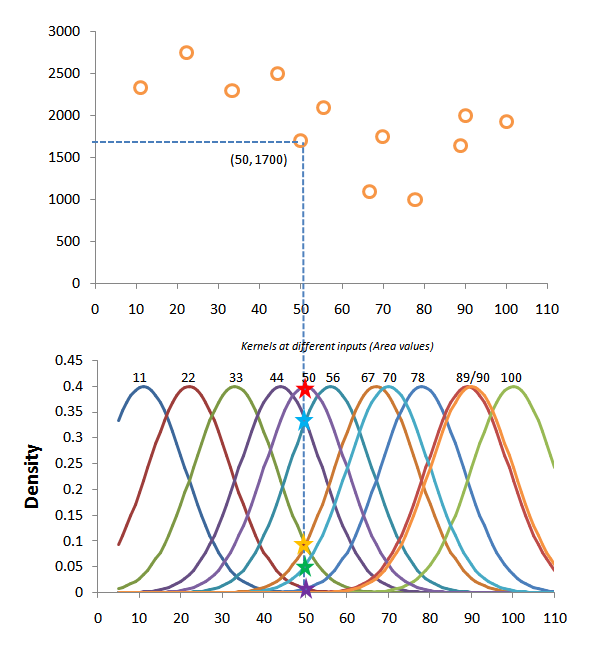
kernel_x = np.arange(min(X)-5,max(X)+5, 0.1)
## Plotting gaussian for all input x points
kernel_fns = {'kernel_x': kernel_x}
for input_x in X:
input_string= 'x_value_{}'.format(np.round(input_x,2))
kernel_fns[input_string] = KernelRegression.kernel_function(bw_manual, kernel_x, input_x)
kernels_df = pd.DataFrame(data=kernel_fns)
y_all = kernels_df.drop(columns='kernel_x')
fig = px.line(kernels_df, x='kernel_x', y=y_all.columns, title='Gaussian for all input points', range_x=[min(X)-5,max(X)+5])
fig.show()widgets.interact(interactive_kernel, bw_manual=dropdown_bw)interactive(children=(Dropdown(description='Szerokość okna', options=(1, 2, 3, 4, 5, 6, 7, 8, 9, 10), value=1)…
<function __main__.interactive_kernel(bw_manual)>
bw_manual = 5
input_x = X[37]
kernel_x = X
weigths = KernelRegression.weights(bw_manual, input_x, X)
# Dataframe for a single observation point x_i. In the code x_i comes from new_x
data = {'Input_x': [input_x for x in kernel_x],
'kernel_x': kernel_x,
'weigth': weigths,
'Y': Y,
'Y=w0*x0+w1*x1+...+w41*x41': ''
}
single_pt_KE = pd.DataFrame(data=data)
single_pt_KE.at[len(Y)//2, 'Y=w0*x0+w1*x1+...+w41*x41'] = KernelRegression.single_y_pred_gauss(3, X[37], X, Y)
single_pt_KE['weigth'] = single_pt_KE['weigth'].map('{:,.10f}'.format)
single_pt_KE['kernel_x'] = single_pt_KE['kernel_x'].map('K({})'.format)
single_pt_KE| Input_x | kernel_x | weigth | Y | Y=w0*x0+w1*x1+...+w41*x41 | |
|---|---|---|---|---|---|
| 0 | 28.6 | K(2.0) | 0.0000001506 | 11 | |
| 1 | 28.6 | K(2.2) | 0.0000001862 | 9 | |
| 2 | 28.6 | K(2.2) | 0.0000001862 | 14 | |
| 3 | 28.6 | K(2.5) | 0.0000002551 | 22 | |
| 4 | 28.6 | K(3.4) | 0.0000006423 | 17 | |
| 5 | 28.6 | K(3.6) | 0.0000007851 | 15 | |
| 6 | 28.6 | K(4.0) | 0.0000011675 | 16 | |
| 7 | 28.6 | K(4.8) | 0.0000025327 | 19 | |
| 8 | 28.6 | K(5.0) | 0.0000030615 | 32 | |
| 9 | 28.6 | K(5.4) | 0.0000044518 | 27 | |
| 10 | 28.6 | K(5.6) | 0.0000053554 | 23 | |
| 11 | 28.6 | K(5.7) | 0.0000058703 | 11 | |
| 12 | 28.6 | K(6.2) | 0.0000092341 | 29 | |
| 13 | 28.6 | K(6.9) | 0.0000171209 | 18 | |
| 14 | 28.6 | K(7.2) | 0.0000221736 | 29 | |
| 15 | 28.6 | K(7.3) | 0.0000241504 | 31 | |
| 16 | 28.6 | K(7.7) | 0.0000338487 | 37 | |
| 17 | 28.6 | K(8.6) | 0.0000706756 | 53 | |
| 18 | 28.6 | K(9.0) | 0.0000970184 | 39 | |
| 19 | 28.6 | K(9.5) | 0.0001428651 | 44 | |
| 20 | 28.6 | K(10.5) | 0.0003006361 | 42 | |
| 21 | 28.6 | K(10.5) | 0.0003006361 | 36 | 32.501314 |
| 22 | 28.6 | K(10.7) | 0.0003471999 | 43 | |
| 23 | 28.6 | K(10.8) | 0.0003728964 | 34 | |
| 24 | 28.6 | K(11.0) | 0.0004296198 | 75 | |
| 25 | 28.6 | K(11.3) | 0.0005296946 | 34 | |
| 26 | 28.6 | K(11.9) | 0.0007965586 | 46 | |
| 27 | 28.6 | K(12.2) | 0.0009715577 | 46 | |
| 28 | 28.6 | K(15.1) | 0.0055032862 | 25 | |
| 29 | 28.6 | K(15.1) | 0.0055032862 | 30 | |
| 30 | 28.6 | K(16.5) | 0.0112700122 | 40 | |
| 31 | 28.6 | K(17.4) | 0.0171422369 | 32 | |
| 32 | 28.6 | K(18.4) | 0.0262993813 | 32 | |
| 33 | 28.6 | K(18.5) | 0.0273891079 | 22 | |
| 34 | 28.6 | K(21.6) | 0.0790709391 | 31 | |
| 35 | 28.6 | K(21.8) | 0.0835583686 | 4 | |
| 36 | 28.6 | K(23.3) | 0.1201265071 | 29 | |
| 37 | 28.6 | K(28.6) | 0.2106810573 | 27 | |
| 38 | 28.6 | K(29.1) | 0.2096302812 | 34 | |
| 39 | 28.6 | K(34.1) | 0.1150475376 | 68 | |
| 40 | 28.6 | K(36.2) | 0.0663633810 | 41 | |
| 41 | 28.6 | K(39.7) | 0.0179240863 | 147 |
from sklearn.linear_model import LinearRegression
# linear regression
reg = LinearRegression().fit(X.reshape(-1, 1), Y.reshape(-1, 1))
Y_pred_linear = reg.predict(X.reshape(-1, 1))
# kernel regression
Y_pred_gauss = KernelRegression.ker_reg(X, Y, bw_manual, 'gauss')
Y_pred_epanechnikov = KernelRegression.ker_reg(X, Y, bw_manual, 'epanechnikov')
fig = px.scatter(x=X,y=Y)
fig.add_trace(go.Scatter(x=X, y=np.array(Y_pred_gauss), name='Gauss', mode='lines'))
fig.add_trace(go.Scatter(x=X, y=np.array(Y_pred_epanechnikov), name='Epanechnikov', mode='lines'))
fig.add_trace(go.Scatter(x=X, y=np.array(Y_pred_linear.flatten().tolist()), name='Linear', mode='lines'))